Úvod
Po celá desetiletí je známo, že interakce mezi potravinami a léčivy (FDI) a interakce mezi bylinami a léčivy omezují úspěšnost léčby. Enormní počet možných interakcí mezi genetickými variacemi, lékařskými režimy a četnými bioaktivními sloučeninami nalezenými v potravinách a bylinách má za následek ohromnou složitost. Moderní nástroje, jako je analýza velkých dat, strojové učení a simulace interakcí protein-ligand, nám mohou pomoci odpovědět na celou řadu otázek: Mohla by volba jídla přispět k selhání terapeutických režimů a pokud ano, jak? Které potraviny by měly být konzumovány před užitím předepsaného léku? A pravděpodobně nejzajímavější otázka: Jak můžeme pomocí těchto nástrojů předpovědět osobní PZI? Je zřejmé, že mnoho odpovědí spočívá v metabolismu léků, potravin a bylin cytochromem P450 3A4 (CYP3A4) v játrech a zažívacím traktu (Galetin et al., 2010; Basheer a Kerem, 2015).
Většina genů kódujících enzymy CYP je polymorfní. Doposud nejkomplexnějším zdrojem informací popisujících alely CYP je Pharmagene Variation Consortium1, ve kterém je zastoupeno méně než 100 alel CYP3A4. Z nich méně než 40 jsou exonické SNP (polymorfismy s jedním nukleotidy), které vedou k modifikované proteinové sekvenci. Malý počet subjektů ve všech dříve publikovaných pracích o mutacích CYP3A4 nám poskytuje omezená data týkající se skutečných frekvencí mutací CYP3A4 v celé populaci a v definovaných skupinách.
Nejen, že spolehlivé informace o výskytu SNP jsou neúplné , také jejich klinické důsledky jsou ve většině případů dosud nejasné (Zanger et al., 2014). Pochopit, které a kdy SNP mohou mít klinický význam, je nesmírně složitý úkol. Testy in vitro jsou časově náročné, nákladné a prakticky málo relevantní vzhledem k velkému množství mutací a nekonečnému počtu kombinací potravin a léků. Metody molekulárního modelování, včetně dokování a výpočtů vazby volné energie, mohou sloužit k předpovědi potenciálních účinků SNP a mnoha sloučenin na metabolismus zprostředkovaný CYP3A4 (Lewis et al., 1998). Například nekovalentní, hydrofobní, elektrostatické a van der Waalsovy interakce přispívají k orientaci sloučeniny, a tedy k její vazbě a reakci na aktivním místě enzymu. Na druhé straně budou určovat afinitu a specificitu enzymu k různým substrátům a účinnost inhibitorů enzymů (Kirchmair et al., 2012; Basheer et al., 2017).
Zde navrhujeme nový přístup k měření alelické frekvence mutací CYP3A4 u různých etnických skupin. Tento komplexní přístup má pravomoc zvýraznit mutace, které převládají u konkrétních etnických skupin, a v kombinaci s screeningem interagujících chemikálií, např. Inhibitory z potravin, umožní objasnění účinků konkrétních mutací na interakci mezi drogami a potravinami a slouží jako počáteční krok k personalizované medicíně a výživě. Tato práce může zvýšit povědomí o možném klinickém významu SNP CYP3A4, které mění bílkoviny, a také navrhuje několik nezbytných nástrojů pro propagaci a aplikaci přesné a personalizované medicíny.
Materiály a metody
Screening a analýza dat
Datová sada variant CYP3A4 byla stažena z prohlížeče gnomAD browser2 jako soubor CVS. Pro analýzu a vizualizaci dat byl použit Python 2.7 s balíčky NumPy, pandy a matplotlib (viz doplňkový datový list S1). Aglomerativní hierarchické shlukování bylo provedeno pomocí softwaru Expander 7 (Shamir et al., 2005) s Pearsonovým korelačním koeficientem jako měřítko podobnosti a typu úplné vazby. Pro seskupení SNP byla stanovena prahová hodnota 0,6.
V modelování polymorfismu in silico
Pro vydání výpočetní modelování. Dokovací model CYP3A4 byl sestaven, jak bylo popsáno dříve (Basheer et al., 2017). Stručně řečeno, krystalová struktura CYP3A4 (položka PDB 2V0M) byla zpracována, upravena a rafinována podle kroků Průvodce přípravou proteinu. Na základě těžiště ketokonazolu v původním vazebném místě v krystalové struktuře byla vytvořena dokovací mřížka s omezením koordinace kovů pro Fe2 + ve skupině hemu. Pro simulace dokování bylo vybráno sedm mutací, jedna jako reprezentant pro každou etnickou skupinu (tabulky 1, 2). Pro každou variantu proteinu byla před kroky přípravy proteinu zavedena jednobodová mutace. 3D struktury ligandů byly generovány na základě 2D struktur z PubChem3 a připraveny pro dokování pomocí úlohy LigPrep.Pro dokovací model byly použity silové pole OPLS3 a výchozí možnosti klouzání pro standardní přesnost, s výjimkou, že bylo použito omezení kovové koordinace, stejně jako 30 pozic pro zahrnutí počtu pozic a 10 pozic pro počet pozic k zápisu ven. Pro každý ligand byl vybrán výsledek ukotvení s nejnižším skóre emodelu Glide.

Tabulka 1. Vybrané reprezentativní SNP pro sedm etnických skupin.

Tabulka 2. Frekvence (%) vybraných mutací podle etnické skupiny.
Výsledky
Databáze agregace genomu (gnomAD; viz poznámka pod čarou 2) agreguje data sekvenování exome i genomu z široké škály rozsáhlých sekvenčních projektů. Zahrnuje údaje ze 125 748 sekvencí exomu a 15 708 sekvencí celého genomu od 141 456 nepříbuzných jedinců představujících sedm etnických populací (Lek et al., 2016). Databáze GnomAD představuje 856 variant CYP3A4, z nichž 397 je intronických a až 459 exonických. Z exonických SNP je 312 missense mutací, což naznačuje, že ovlivňují proteinovou strukturu. Gen CYP3A4 je dlouhý 34 205 bp. Jeho 13 exonů obsahuje oblast kódující 1512 bp, která produkuje protein 504 aminokyselin. 412 exonických SNP s jedinečnými pozicemi v tomto genu vede k exonické hustotě SNP 272 / kbp (doplňková tabulka S1).
Výpočet diferenciálních frekvencí alel na etnickou skupinu ukazuje, že některé populace vykazují vyšší frekvence mutací (Obrázek 1A). Většina mutací CYP3A4 v evropské populaci je skutečně vzácná, jak se běžně předpokládá, zatímco mutace v jiných populacích, jako je africká a východní Asie, jsou mnohem častější (doplňková tabulka S2).
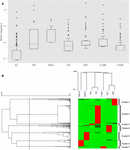
Obrázek 1. Analýza SNP missense CYP3A4 v sedmi odlišných populacích. (A) Krabicový graf alelických frekvencí v měřítku protokolu. Krabice představují mezikvartilní rozsah (IQR), modré čáry představují mediány, vousy představují data do 1,5 IQR a odlehlé hodnoty jsou zobrazeny jako malé kroužky. (B) Hierarchické shlukování alelických frekvencí. Každý řádek představuje jeden SNP. Každý sloupec představuje odlišnou etnickou populaci. Frekvence alel SNP v každé z populací je reprezentována barvou odpovídající buňky v souboru matice. Zelená a červená představují nízkou a vysokou frekvenci. Horní dendrogram ukazuje podobnosti ve vzorci frekvencí alel mezi každou skupinou subjektů. Levý dendrogram představuje shlukování genů ve dvou skupinách. Přerušovaná čára představuje prahovou hodnotu vzdálenosti 0,6 použitou pro rozdělení do skupin. EU – evropský (ne finský; n = 64 603), FIN – evropský (finský; n = 12 562), ASH J – aškenázský židovský (n = 5 185), LTN – latino (n = 17 720), AFR – africký (n = 12 487), E ASN – východní Asie (n = 9 977), S ASN – jižní Asie (n = 64 603).
K seskupení variant s podobnými frekvenčními vzory jsme použili hierarchické shlukování. Naše analýza dat poskytla sedm odlišných shluků (obrázek 1B). Dále je jasně pozorováno, že vysokofrekvenční SNP v každém klastru jsou charakteristické pro jednu konkrétní populaci. Hierarchická shluková analýza etnických skupin podporuje asociaci mezi genetickou rozptylem a etnickým původem seskupením příbuzných etnických skupin, jako jsou jižní a východní Asiaté, stejně jako finští a nefinští Evropané.
K posouzení byl použit výpočetní model možný vliv bodových mutací v CYP3A4 na jeho schopnost vázat substráty a inhibitory. CYP3A4 je schopen oxidovat širokou škálu endogenních a xenobiotických sloučenin. Zde byl vybrán ketokonazol jako reprezentativní léčivo a velmi účinný specifický inhibitor; androstendion a testosteron byly vybrány jako reprezentativní endogenní hormon; a demethoxykurkumin a epigalokatechin byly vybrány jako zástupci dietních bioaktivů. Byl vytvořen dokovací model pro predikci vazebných pozic vybraných sloučenin ve vazebném místě CYP3A4. Model byl nejprve ověřen úspěšným obnovením pólu ketokonazolu ve vazebném místě s RMSD 1,52 Á vzhledem k původní krystalové struktuře. Sedm mutantních proteinů bylo navrženo na základě krystalové struktury proteinu divokého typu (doplňkový obrázek S1). Pro každou etnickou skupinu byla jako reprezentativní vybrána nejčastější jedinečná mutace. Účinek jednotlivých mutací na vazbu substrátu byl hodnocen na základě srovnání mezi dokovacími pozicemi na nativním proteinu a na variantních proteinech. Změny v dokovacích pozicích, pokud jde o RMSD, jsou shrnuty v tabulce 3.

Tabulka 3. RMSD vzhledem k WT dokovacích ligandů k vazebným místům sedmi variant CYP3A4.
Bylo zjištěno, že účinek CYP3A4 SNP na vazbu substrátu je specifický pro mutační substrát. Pouze v několika případech mutace způsobily změnu vazebné pozice ligandu ve vazebné kapse. Dokovací pozice testosteronu byla ve všech sedmi testovaných variantách stejná. Varianty E262K, D174H a K168N nezpůsobily změnu vazebné pózy v žádné z testovaných molekul. Mutace L373F a T163A však změnily vazebnou pózu androstendionu tak, že byl umístěn spíše paralelně s hemovou skupinou než kolmo k ní, jako v proteinu WT. Androstendion byl také rotován tak, aby se cyklopentanonová skupina nacházela proximálně k hemu, místo cyklohexanonové skupiny v proteinu WT. Mutace S222P a L293P způsobily pouze malou rotaci vazebné pozice androstendionu (obrázek 2A). Ze všech zkoumaných mutací pouze S222P způsobil podstatné změny v dokovacích pozicích ketokonazolu a demethoxykurkuminu ve vazebném místě (obrázky 2B, C); vzhledem k tomu, že pro epigalokatechin byla mutací měnící pózy L373F (obrázek 2D).
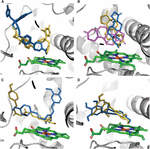
Obrázek 2. Modely ligandů ukotvených ve vazebném místě CYP3A4. (A) Ketokonazol, (B) androstendion, (C) demethoxykurkumin a (D) epigalokatechin. Místo vázající protein je představováno šedými stužkami; hém je reprezentován zelenými tyčinkami, dokovací pózy v proteinu WT a u mutantů S222P a L373F jsou zobrazeny jako oranžové, modré a fialové tyčinky. Dokovací pozice Androstendionu ve variantách L293P a T136A se překrývají s pozicemi ve variantách S222P a L373F.
Diskuse
Cytochrom P450 3A4 je hlavní enzym odpovědný za interakce mezi potravinami a léky. Současný výzkum mutací v CYP3A4 byl zaměřen na několik desítek SNP nalezených v určených studiích (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. , 2002; Murayama a kol., 2002). Jak je zde prokázáno, představují špičku ledovce s ohledem na prevalenci a potenciální výsledky mutací CYP3A4. Hojnost projektů sekvenování velkého genomu a exome otevřela novou cestu k identifikaci mnoha neznámých mutací. Zde ukážeme, že dříve prezentované mutace jsou pouze špičkou ledovce, demonstrací 856 mutací existujících v CYP3A4, z nichž jedna třetina modifikuje proteinovou strukturu. Pomocí kohorty 141 456 nepříbuzných jedinců byly vypočteny přesné alelické frekvence mutací CYP3A4 pro sedm samostatných etnik. Pokud je nám známo, jedná se o dosud největší a nejkomplexnější studii rozsáhlých dat exonových mutací CYP3A4 a jejich alelických frekvencí v různých populacích.
Polymorfní enzymy CYP3A4 mohou být při vysvětlování velmi důležité rozdíly v účinnosti a toxicitě léku u různých jedinců. Mutace v genu CYP3A4 mohou vést ke zrušení, snížení, změně nebo zvýšení enzymatické aktivity. Exonové mutace mohou modifikovat enzymatickou aktivitu, jak bylo prokázáno v několika klinických studiích s vybranými substráty. Některé případy změněného metabolismu v důsledku SNP v CYP3A4 již byly popsány v literatuře (Eiselt et al., 2001; Miyazaki et al., 2008). Navzdory funkčnímu a klinickému významu SNP v CYP3A4 a pravděpodobně kvůli jejich relativně nízké identifikované frekvenci v obecné populaci se polymorfismu v CYP3A4 nedostala pozornost, kterou si zaslouží.
Zde předpovědělo sedm mutací účinek SNP na orientaci vazby na substrát a inhibitor. V literatuře polymorfismus CYP3A4 rozděluje obecnou populaci do tří skupin – pomalí metabolizátoři, normální metabolizátoři a rychlí metabolizátoři, založené na intronních SNP, které spíše než strukturu modifikují úrovně exprese (Zanger a Schwab, 2013). Naše výpočty naznačují další klasifikaci: změněné metabolizátory. Některé mutace navržené naším virtuálním modelem by způsobily změnu vazebné orientace jednotlivých ligandů. Očekává se, že tyto změny sníží pravděpodobnost enzymatické oxidace v důsledku větší vzdálenosti od hemu nebo povedou k produktům, které by jinak nebyly zřejmé během testů toxicity prováděných v rámci procesu vývoje léčiva. Jak však náš model předpovídá, pro většinu substrátů jsou mutace CYP3A4 benigní.
Upravená poloha substrátu ve vazebné kapse v důsledku strukturální změny proteinu je pouze jedním možným mechanismem, kterým by mutace mohla změnit aktivitu proteinu . Narušené ukotvení proteinu k membráně, poškození kanálů vedoucích substrátem a kompromitovaný výstup produktů představují další mechanismy mutační změny aktivity proteinu. Jak je zde ukázáno, účinek každé mutace je specifický pro daný substrát.Stanovení kombinací substrátů a mutací, které by mohly upravit enzymatickou aktivitu pomocí tradičních metod in vitro, je pracné, což zdůrazňuje potřebu prediktivních virtuálních nástrojů při řešení této složité hádanky. rychle roste. Predikce modifikovaného metabolismu léčiv na základě individuálního polymorfismu v CYP3A4 se zdá být jen otázkou času. Zde navrhujeme, aby odlišné etnické skupiny nesly jedinečné sady SNP CYP3A4. Etnická příslušnost může skutečně sloužit jako první proveditelný krok v personalizované medicíně před zavedením individuálního screeningu DNA pro všechny. Zajímavé je, že etnický původ má ještě jeden důsledek pro metabolismus léčiv CYP3A4, který je hlavním faktorem při určování volby potravin a stravovacích návyků. Lze navrhnout, že terapeutické režimy by měly být specificky navrženy pro každou etnickou skupinu, alespoň pro léky, které jsou vysoce metabolizovány CYP3A4. To zdůrazňuje příležitosti k využití a integraci databází a hlubokého učení k identifikaci toho, jak SNP, etnický původ, dietní sloučeniny a léky mění aktivitu CYP3A4 a úspěch lékařského režimu.
Dostupnost dat
V této studii byly analyzovány veřejně dostupné datové soubory. Tyto údaje najdete zde: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Příspěvky autorů
Všichni uvedení autoři významně, přímo a intelektuálně přispěli k práce a schválili jej ke zveřejnění.
Prohlášení o střetu zájmů
Autoři prohlašují, že výzkum byl proveden bez jakýchkoli obchodních nebo finančních vztahů, které by bylo možné považovat za potenciální střet zájmů.
Doplňkový materiál
OBRÁZEK S1 | 3D pásový model CYP3A4 a umístění mutovaných aminokyselin v sedmi variantních proteinech určených pro dokování. Hem je reprezentován jako zelené tyčinky, Fe2 + je reprezentován jako červená koule, SNP použité v analýze in silico jsou reprezentovány jako červené oblasti na pásu a R skupiny mutovaných aminokyselin ve variantních modelech jsou zobrazeny výslovně jako světle šedé tyčinky.
TABULKA S1 | Typy CYP3A4 SNP v populaci 141 456 nepříbuzných jedinců představujících 7 etnických populací.
TABULKA S2 | CYP3A4 SNP podle etnické skupiny.
Poznámky
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov