Johdanto
Vuosikymmenien ajan ruoka-lääke-yhteisvaikutusten ja yrtti-lääke-vuorovaikutusten tiedetään rajoittavan lääkehoitojen onnistumista. Geenimuunnelmien, lääketieteellisten järjestelmien ja elintarvikkeissa ja yrtteissä olevien lukemattomien bioaktiivisten yhdisteiden välinen valtava määrä mahdollisia vuorovaikutuksia johtaa ylivoimaisesti monimutkaisuuteen. Nykyaikaiset työkalut, kuten isojen tietojen analysointi, koneoppiminen ja proteiini-ligandi-vuorovaikutuksen simulointi, voivat auttaa meitä vastaamaan useisiin kysymyksiin: Voisiko ruokavalinnat myötävaikuttaa terapeuttisten järjestelmien epäonnistumiseen ja jos on, niin miten? Mitä ruokia tulisi syödä ennen määrätyn lääkkeen ottamista? Ja luultavasti mielenkiintoisin kysymys: Kuinka voimme käyttää näitä työkaluja henkilökohtaisten suorien sijoitusten ennustamiseen? On selvää, että monet vastaukset liittyvät lääkkeiden, elintarvikkeiden ja yrttien aineenvaihduntaan sytokromi P450 3A4: n (CYP3A4) kautta maksassa ja ruoansulatuskanavassa (Galetin ym., 2010; Basheer ja Kerem, 2015).
Suurin osa CYP-entsyymejä koodaavista geeneistä on polymorfisia. Tähän mennessä kattavin tietolähde, joka kuvaa yksityiskohtaisesti CYP-alleeleja, on Pharmacogene Variation Consortium1, jossa on edustettuna alle 100 CYP3A4-alleelia. Näistä alle 40 on eksonisia SNP: itä (yhden nukleotidin polymorfismeja), jotka johtavat modifioituun proteiinisekvenssiin. Pieni tutkimushenkilöiden määrä kaikissa aiemmin julkaistuissa CYP3A4-mutaatioita käsittelevissä teoksissa antaa meille rajallista tietoa CYP3A4-mutaatioiden todellisista esiintymistiheyksistä koko populaatiossa ja määritellyissä ryhmissä.
Paitsi että luotettava tieto SNP: n esiintyvyydestä on puutteellista , myös niiden kliiniset vaikutukset ovat vielä epäselvät useimmissa tapauksissa (Zanger et al., 2014). Ymmärtäminen, millä ja milloin SNP: llä voi olla kliininen merkitys, on erittäin monimutkainen tehtävä. In vitro -määritykset ovat aikaa vieviä, kalliita ja käytännöllisesti katsoen vähäisiä, kun otetaan huomioon mutaatioiden suuri määrä ja loputon määrä ruoka-lääke-yhdistelmiä. Molekyylimallinnusmenetelmät, mukaan lukien telakointi ja vapaan energian sitoutumislaskelmat, voivat palvella ennustamaan SNP: n ja monien yhdisteiden mahdollisia vaikutuksia CYP3A4-välitteiseen metaboliaan (Lewis et ai., 1998). Esimerkiksi ei-kovalenttiset, hydrofobiset, sähköstaattiset ja van der Waalsin vuorovaikutukset edistävät kaikki yhdisteen orientoitumista ja siten sen sitoutumista ja reaktiota entsyymin aktiivisessa kohdassa. Ne puolestaan määrittelevät entsyymin affiniteetin ja spesifisyyden eri substraatteihin sekä entsyymi-inhibiittoreiden voimakkuuden (Kirchmair et al., 2012; Basheer et al., 2017).
Tässä ehdotamme uutta lähestymistapa CYP3A4-mutaatioiden alleelitiheyden mittaamiseen eri etnisissä ryhmissä. Tällä kokonaisvaltaisella lähestymistavalla on valta tuoda esiin mutaatiot, jotka ovat yleisiä tietyissä etnisissä ryhmissä, ja yhdistettynä vuorovaikutuksessa olevien kemikaalien seulontaan, esim. Ruoasta saatavat estäjät, mahdollistavat tiettyjen mutaatioiden vaikutusten selvittämisen huumeiden ja elintarvikkeiden vuorovaikutuksessa. askel kohti henkilökohtaista lääketiedettä ja ravintoa. Tämä työ voi lisätä tietoisuutta proteiineja muuttavien CYP3A4 SNP: ien mahdollisesta kliinisestä merkityksestä ja ehdottaa myös muutamia tarvittavia työkaluja tarkkuus- ja yksilöllisen lääketieteen edistämiseen ja soveltamiseen.
Materiaalit ja menetelmät
Tietokannan seulonta ja tietojen analysointi
CYP3A4-varianttien tietojoukko ladattiin gnomAD-selaimelta2 CVS-tiedostona. Datan analysointiin ja visualisointiin käytettiin Python 2.7 -pakettia, jossa oli NumPy, pandas ja matplotlib (katso Supplementary Data Sheet S1). Agglomeratiivinen hierarkkinen klusterointi suoritettiin käyttämällä Expander 7 -ohjelmistoa (Shamir et ai., 2005) Pearsonin sijoituskorelointikertoimen kanssa samankaltaisuuksien ja täydellisen linkitystyypin mittana. SNP: n ryhmittelylle asetettiin etäisyysraja 0,6.
In silico Polymorphism Modeling
Maestro 2017-2 -julkaisua (Schrodinger, New York, NY, Yhdysvallat) käytettiin laskennallinen mallinnus. CYP3A4-telakointimalli rakennettiin aiemmin kuvatulla tavalla (Basheer et al., 2017). Lyhyesti sanottuna CYP3A4-kiteiden rakenne (PDB-merkintä 2V0M) käsiteltiin, modifioitiin ja puhdistettiin ohjatun proteiinin valmisteluvaiheen vaiheiden mukaisesti. Telakointiristikko, jossa oli hemiryhmän Fe2 +: n metallikoordinaatiorajoitus, muodostettiin ketokonatsolin sentroidin perusteella alkuperäisessä sidoskohdassa kristallirakenteessa. Telakointisimulaatioihin valittiin seitsemän mutaatiota, yksi edustaja kullekin etniselle ryhmälle (taulukot 1, 2). Kutakin proteiinimuunnosta varten lisättiin yhden pisteen mutaatio ennen proteiinin valmistusvaiheita. Ligandien 3D-rakenteet kehitettiin PubChem3: n 2D-rakenteiden perusteella ja valmisteltiin telakointia varten käyttämällä LigPrep-tehtävää.Telakointimallissa käytettiin OPLS3-voimakenttää ja vakiotarkkuuden oletusliukuvaihtoehtoja lukuun ottamatta sitä, että käytettiin metallikoordinaatiorajoitusta, samoin kuin 30 asentoa sisällytettävien poseerien lukumäärälle ja 10 asentoa kirjoitettavien poseerien lukumäärälle. ulos. Jokaiselle ligandille valittiin telakointitulos, jolla oli pienin Glide-mallin pisteet.

Taulukko 1. Valitut edustavat SNP: t seitsemälle etniselle ryhmälle.

Taulukko 2. Valittujen mutaatioiden taajuus (%) etnisten ryhmien mukaan.
Tulokset
Genomien aggregaatiotietokanta (gnomAD; katso teksti alaviite 2) yhdistää sekä exome- että genomisekvensointitiedot useista laajamittaisista sekvensointiprojekteista. Se sisältää tietoja 125 748 eksomisekvenssistä ja 15 708 kokonaisen genomin sekvenssiä 141 456 etuyhteydettömästä yksilöstä, jotka edustavat seitsemää etnistä populaatiota (Lek et ai., 2016). GnomAD-tietokanta esittää 856 CYP3A4-muunnosta, joista 397 ovat intronisia ja jopa 459 eksonisia. Eksonisista SNP: stä 312 on missense-mutaatioita, mikä osoittaa, että ne vaikuttavat proteiinirakenteeseen. CYP3A4-geeni on 34205 emäsparia pitkä. Sen 13 eksonia käsittää 1 512 emäsparin koodaavan alueen, joka tuottaa 504 aminohapon proteiinia. 412 eksonista SNP: tä, joilla on ainutlaatuiset sijainnit tässä geenissä, johtavat eksonin SNP-tiheyteen 272 / kbp (täydentävä taulukko S1).
Eri alleelitaajuuksien laskeminen etnistä ryhmää kohden paljastaa, että joillakin populaatioilla on korkeammat mutaatiotaajuudet. (Kuva 1A). Suurin osa Euroopan väestön CYP3A4-mutaatioista on todellakin harvinaisia, kuten yleisesti ajatellaan, kun taas muiden populaatioiden, kuten Afrikan ja Itä-Aasian, mutaatiot ovat paljon yleisempiä (täydentävä taulukko S2).
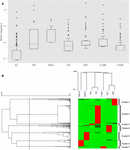
Kuva 1. Analyysi CYP3A4-missense-SNP: stä seitsemässä erillisessä populaatiossa. (A) Alleelitaajuuksien lokimittakaavan laatikkokaavio. Laatikot edustavat kvartiilien välistä aluetta (IQR), siniset viivat edustavat mediaaneja, viikset edustavat tietoja 1,5 IQR: n sisällä ja poikkeamat esitetään pieninä ympyröinä. (B) Alleelisten taajuuksien hierarkkinen ryhmittely. Jokainen rivi edustaa yhtä SNP: tä. Jokainen sarake edustaa erillistä etnistä väestöä. SNP: iden alleelitaajuutta kussakin populaatiossa edustaa matriisitiedoston vastaavan solun väri. Vihreä ja punainen edustavat vastaavasti matalaa ja korkeaa taajuutta. Ylempi dendrogrammi näyttää samanlaisia alleelitaajuuskuviossa kunkin tutkimusryhmän välillä. Vasen dendrogrammi edustaa geenien ryhmittymistä kahteen ryhmään. Katkoviiva edustaa 0,6 etäisyyden kynnystä, jota käytetään ryhmiin jakamiseen. EU – eurooppalainen (ei-suomalainen; n = 64603), FIN – eurooppalainen (suomi; n = 12 562), ASH J – Ashkenazi-juutalainen (n = 5185), LTN – latino (n = 17 720), AFR – afrikkalainen (n = 12487), E ASN – itäaasialainen (n = 9977), S ASN – eteläaasialainen (n = 64603).
Käytimme hierarkkista klusterointia ryhmittelemään variantteja, joilla on samanlainen taajuusmalli. Tietoanalyysimme tuotti seitsemän erillistä klusteria (kuva 1B). Lisäksi on selvästi havaittu, että jokaisen klusterin suurtaajuiset SNP: t ovat ominaisia yhdelle tietylle populaatiolle. Etnisten ryhmien hierarkkinen ryhmittelyanalyysi tukee geneettisen varianssin ja etnisyyden välistä yhteyttä ryhmittelemällä yhteen etnisiin ryhmiin kuuluvat etelä- ja itäaasialaiset sekä suomalaiset ja suomalaiset eurooppalaiset.
Laskennallista mallia käytettiin arvioitaessa CYP3A4: n pistemutaatioiden mahdollinen vaikutus sen kykyyn sitoa substraatteja ja estäjiä. CYP3A4 pystyy hapettamaan laajan valikoiman endogeenisiä ja ksenobioottisia yhdisteitä. Tässä ketokonatsoli valittiin edustavaksi lääkkeeksi ja erittäin tehokkaaksi spesifiseksi inhibiittoriksi; androstenedioni ja testosteroni valittiin edustavaksi endogeeniseksi hormoniksi; ja demetoksikurkumiini ja epigallokatekiini valittiin ruokavalion bioaktiivisten aineiden edustajiksi. Telakointimalli rakennettiin ennustamaan valittujen yhdisteiden sitoutumisasennot CYP3A4: n sitoutumiskohdassa. Malli validoitiin ensin palauttamalla onnistuneesti ketokonatsoliasema sitoutumiskohdassa RMSD: n ollessa 1,52 Å alkuperäisen kiderakenteen suhteen. Seitsemän mutanttiproteiinia suunniteltiin villityypin proteiinin kiderakenteen perusteella (täydentävä kuva S1). Jokaiselle etniselle ryhmälle valittiin tyypillisin ainutlaatuinen mutaatio edustavaksi. Yksittäisten mutaatioiden vaikutus substraatin sitoutumiseen arvioitiin telakointiasemien natiiviin proteiiniin ja varianttiproteiineihin vertailun perusteella. Telakointiasemien muutokset RMSD: n suhteen on esitetty yhteenvetona taulukossa 3.

Taulukko 3. Telakointiligandien RMSD suhteessa WT: hen seitsemän CYP3A4-variantin sitoutumiskohtiin.
CYP3A4 SNP: n vaikutuksen substraatin sitoutumiseen todettiin olevan mutaatio-substraattispesifinen. Vain muutamissa tapauksissa mutaatiot aiheuttivat muutoksen sitoutumistaskussa olevan ligandin sitoutumisasennossa. Testosteronin telakointiasema oli sama kaikissa seitsemässä testatussa variantissa. E262K-, D174H- ja K168N-variantit eivät aiheuttaneet sitoutumisasennon muutosta missään testatussa molekyylissä. L373F- ja T163A-mutaatiot kuitenkin muuttivat androsteenidionin sitoutumisasennon siten, että se sijaitsi yhdensuuntaisesti hemiryhmän kanssa eikä sen suhteen kohtisuorassa, kuten WT-proteiinissa. Androsteenidionia kierrettiin myös siten, että syklopentanoniryhmä sijaitsee hemin läheisyydessä WT-proteiinin sykloheksanoniryhmän sijasta. S222P- ja L293P-mutaatiot aiheuttivat vain pienen rotaation androstenedionin sitoutumisasennossa (kuvio 2A). Kaikista tutkituista mutaatioista vain S222P aiheutti merkittäviä muutoksia ketokonatsolin ja demetoksikurkumiinin telakointiasemissa sitoutumiskohdassa (kuviot 2B, C); kun taas epigallokatekiinille poseja muuttava mutaatio oli L373F (kuva 2D).
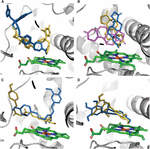
Kuva 2. CYP3A4: n sitoutumiskohtaan telakoitujen ligandien mallit. (A) ketokonatsoli, (B) androsteenidioni, (C) demetoksikurkumiini ja (D) epigallokatekiini. Proteiinia sitovaa kohtaa edustavat harmaat nauhat; hemiä edustavat vihreät sauvat, telakointiasennot WT-proteiinissa ja S222P- ja L373F-mutantit esitetään vastaavasti oransseina, sinisinä ja violetteina. Androstenedionin telakointiasennot L293P- ja T136A-muunnoksissa ovat päällekkäisiä vastaavien S222P- ja L373F-varianttien kanssa.
Keskustelu
Sytokromi P450 3A4 on tärkein entsyymi, joka on vastuussa elintarvikkeiden ja lääkkeiden yhteisvaikutuksista. Nykyinen tutkimus CYP3A4: n mutaatioista on keskittynyt muutamaan tusinaan nimetyissä tutkimuksissa löydettyjä SNP: itä (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. (2002; Murayama et ai., 2002). Kuten täällä on osoitettu, ne edustavat jäävuoren kärkeä ottaen huomioon CYP3A4-mutaatioiden esiintyvyyden ja mahdolliset tulokset. Suuren genomin ja eksomin sekvensointiprojektien runsaus on avannut uuden tavan tunnettujen mutaatioiden tunnistamiseen. Tässä osoitetaan, että aiemmin esitetyt mutaatiot ovat vain jäävuoren huippu osoittamalla 856 CYP3A4: ssä esiintyvää mutaatiota, joista kolmasosa modifioi proteiinirakennetta. Käyttämällä 141 456 etuyhteydettömän henkilön kohorttia laskettiin tarkat CYP3A4-mutaatioiden alleelitaajuudet seitsemälle erilliselle etniselle ryhmälle. Parhaan tietomme mukaan tämä on tähän mennessä julkaistu suurin ja kattavin laaja-alainen tutkimus CYP3A4-eksonimutaatioista ja niiden alleelitaajuuksista eri populaatioissa.
Polymorfiset CYP3A4-entsyymit voivat olla erittäin tärkeitä selitettäessä. lääkkeiden tehokkuuden ja toksisuuden erot eri yksilöiden välillä. CYP3A4-geenin mutaatiot saattavat johtaa poistuneeseen, vähentyneeseen, muuttuneeseen tai lisääntyneeseen entsymaattiseen aktiivisuuteen. Eksoniset mutaatiot voivat muuttaa entsymaattista aktiivisuutta, kuten on osoitettu muutamissa kliinisissä tutkimuksissa valituilla substraateilla. Joitakin tapauksia SNP: iden aiheuttamasta metaboliasta CYP3A4: ssä on jo kuvattu kirjallisuudessa (Eiselt et al., 2001; Miyazaki et al., 2008). Huolimatta SNP: iden toiminnallisesta merkityksestä ja kliinisestä merkityksestä CYP3A4: ssä ja mahdollisesti niiden suhteellisen alhaisen tunnistetun esiintyvyyden vuoksi yleisessä populaatiossa, CYP3A4: n polymorfismi ei ole saanut ansaitsemaansa huomiota.
Tässä seitsemän mutaatiota ennusti. SNP: iden vaikutus substraatin ja inhibiittorin sitoutumiseen. Kirjallisuudessa CYP3A4-polymorfismi jakaa väestön kolmeen ryhmään – heikot metaboloijat, normaalit metaboloijat ja nopeat metaboloijat, jotka perustuvat intronisiin SNP: iin, jotka muuttavat ilmentymistasoja rakenteen sijaan (Zanger ja Schwab, 2013). Laskelmissamme ehdotetaan lisäluokitusta: muuttuneet metaboloijat. Jotkut virtuaalimallimme ehdottamat mutaatiot aiheuttaisivat muutoksen yksittäisten ligandien sitoutumisorientaatiossa. Näiden muutosten odotetaan vähentävän entsymaattisen hapettumisen todennäköisyyttä, joka johtuu etäisyydestä hemistä, tai johtavat tuotteisiin, jotka muuten eivät olisi ilmeisiä myrkyllisyystestien yhteydessä osana lääkekehitysprosessia. Kuten mallimme ennustaa, useimmille substraateille CYP3A4-mutaatiot ovat hyvänlaatuisia.
Substraatin modifioitu sijainti sitoutumistaskussa proteiinin rakennemuutoksen vuoksi on vain yksi mahdollinen mekanismi, jolla mutaatio voi muuttaa proteiinin aktiivisuutta . Heikentynyt proteiinin ankkurointi membraaniin, vaurioituneet substraatin johtavat kanavat ja vaurioitunut tuotteiden poistuminen tarjoavat lisämekanismeja mutaatiomuutokselle proteiinin aktiivisuudessa. Kuten tässä on esitetty, jokaisen mutaation vaikutus on substraattikohtainen.Perinteisten in vitro -menetelmien käyttäminen on työlästä selvittää, mitkä substraattien ja mutaatioiden yhdistelmät saattavat muuttaa entsymaattista aktiivisuutta, ja painottaa ennustavien virtuaalisten työkalujen tarvetta tämän monimutkaisen pulman ratkaisemisessa.
Julkinen ja ammatillinen kiinnostus henkilökohtaiseen ja tarkkaan lääketieteeseen kasvaa nopeasti. CYP3A4: n yksilölliseen polymorfismiin perustuvan modifioidun lääkeaineenvaihdunnan ennustaminen näyttää olevan vain ajan kysymys. Tässä ehdotamme, että erillisillä etnisillä ryhmillä olisi ainutlaatuiset joukot CYP3A4 SNP: itä. Etnisyys voi todellakin toimia ensimmäisenä toteuttamiskelpoisena askeleena henkilökohtaisessa lääketieteessä ennen yksilöllisen DNA-näytön käyttöönottoa kaikille. Mielenkiintoista on, että etnisellä alkuperällä on vielä yksi vaikutus CYP3A4-lääkeaineenvaihduntaan, mikä on tärkeä tekijä ruokavalintojen ja ruokailutottumusten määrittämisessä. Voidaan ehdottaa, että terapeuttiset hoidot tulisi suunnitella erikseen kullekin etniselle ryhmälle, ainakin lääkkeille, jotka metaboloituvat voimakkaasti CYP3A4: n kautta. Tämä korostaa mahdollisuuksia hyödyntää ja integroida tietokantoja ja syvällistä oppimista sen selvittämiseksi, kuinka SNP: t, etnisyys, ruokavalion yhdisteet ja lääkkeet muuttavat CYP3A4-aktiivisuutta ja lääketieteellisen järjestelmän onnistumista.
Tietojen saatavuus
Tässä tutkimuksessa analysoitiin julkisesti saatavilla olevia aineistoja. Nämä tiedot ovat täällä: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Kirjoittajan panokset
Kaikki luetellut kirjoittajat ovat antaneet merkittävän, suoran ja älyllisen panoksen teoksen ja hyväksyi sen julkaistavaksi.
Eturistiriita-lausunto
Kirjoittajat vakuuttavat, että tutkimus tehtiin ilman kaupallisia tai taloudellisia suhteita, joita voitaisiin tulkita mahdollisiksi eturistiriita.
Lisämateriaali
KUVA S1 | CYP3A4: n 3D-nauhamalli ja mutatoitujen aminohappojen sijainti seitsemässä telakointiin suunnitellussa proteiiniproteiinissa. Heemi on esitetty vihreinä sauvoina, Fe2 + on esitetty punaisena pallona, in silico -analyysissä käytetyt SNP: t ovat punaisina alueina nauhassa ja muunnettujen aminohappojen R-ryhmät varianttimalleissa on nimenomaisesti vaaleanharmaat sauvat.
TAULUKKO S1 | CYP3A4 SNP-tyypit 141, 456 etuyhteydettömässä yksilössä, jotka edustavat 7 etnistä populaatiota.
TAULUKKO S2 | CYP3A4-SNP: t etnisten ryhmien mukaan.
Alaviitteet
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov