この手法は1980年代後半に開発されたもので、転座(染色体間の再配列)を検出します。
FISHの開発には、各ヒト染色体を分離する必要がありました。続いて、これらの染色体からのDNAが断片化され、細菌細胞に入れられて増幅されました(多くのコピーが生成されます)。このようにして、各染色体から多数のDNAコピーを取得できます。
これらの増幅されたDNAフラグメントは、適切な蛍光(発光)色素で標識され、中期染色体にハイブリダイズ(付着)します。 。蛍光標識されたDNAは、それらが由来する類似の染色体に付着します。 (同じ塩基配列を持つDNA断片は、互いに付着するという特徴があります。)
このようにして、塗装された染色体の一部(たとえば黄色)が別の非染色体と交換された場合-塗装された染色体(赤く染色)、異常な染色体には黄色と赤の両方のセグメントが含まれているため、異常(相互転座と呼ばれる)を検出することができます。通常、2つの染色体は通常DNAの一部を交換するため、2色の染色体のペアを1つの中期で検出できます。
染色体のセット全体を1つで単純に染色するだけでは、相互転座を検出することは困難です。ギムザなどの素材。たとえば、交換された2つのセグメントの長さが同じである場合を想像してみてください。 2つの転座した染色体は、形状と長さの両方で完全に正常に見えるはずです。ただし、FISHを使用すると、そのような転座を明確に検出できます。
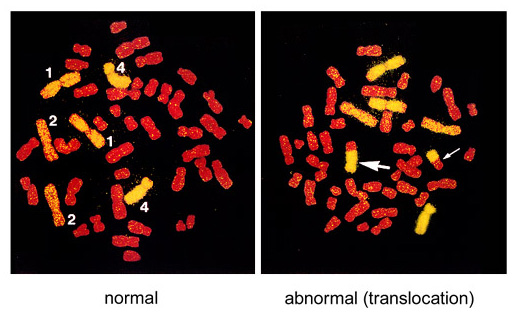
図。 FISHで処理された中期染色体の例
ここでは、染色体1、2、および4がFISHで黄色にラベル付けされ、他の染色体は赤に染色されています。黄色と赤の染色体間の転座が検出されます。左の写真は正常な細胞を表しており(図の数字は染色体番号を示しています)、右の写真は2つの2色染色体(2つの矢印で示されています)による相互転座の例です。