Introduksjon
I flere tiår har mat – medikamentinteraksjoner (FDI) og urt – medikamentinteraksjoner vært kjent for å begrense suksessen til medisinske behandlinger. Det enorme antallet mulige interaksjoner mellom genetiske variasjoner, medisinske regimer og de mange bioaktive forbindelsene som finnes i mat og urter, resulterer i overveldende kompleksitet. Moderne verktøy som big-data analyse, maskinlæring og simulering av protein-ligand interaksjoner kan hjelpe oss med å svare på et helt sett med spørsmål: Kan matvalg bidra til at terapeutiske regimer mislykkes, og i så fall hvordan? Hvilken mat (e) bør konsumeres før du tar et foreskrevet legemiddel? Og sannsynligvis det mest spennende spørsmålet: Hvordan kan vi bruke disse verktøyene til å forutsi personlig FDI? Det er tydelig at mange svar ligger i stoffskiftet av medikamenter, matvarer og urter fra cytokrom P450 3A4 (CYP3A4) i leveren og fordøyelseskanalen (Galetin et al., 2010; Basheer og Kerem, 2015).
Flertallet av gener som koder for CYP-enzymer er polymorfe. Til dags dato er den mest omfattende informasjonskilden som beskriver CYP-alleler Pharmacogene Variation Consortium1, der færre enn 100 alleler av CYP3A4 er representert. Av disse er færre enn 40 eksoniske SNPer (enkle nukleotidpolymorfier) som resulterer i en modifisert proteinsekvens. Det lille antallet fag i alle tidligere publiserte arbeider med CYP3A4-mutasjoner gir oss begrensede data angående sanne frekvenser av CYP3A4-mutasjoner i hele befolkningen og i definerte grupper.
Ikke bare at pålitelig informasjon om SNPs-forekomst er ufullstendig , også er deres kliniske implikasjoner uklare i de fleste tilfeller (Zanger et al., 2014). Å forstå hvilke og når SNP kan ha klinisk betydning er en enormt kompleks oppgave. In vitro-analyser er tidkrevende, dyre og praktisk talt av lav relevans med tanke på den store mengden mutasjoner og det uendelige antallet mat-medikamentkombinasjoner. Molekylære modelleringsmetoder, inkludert forankring og beregning av fri energi bindende, kan tjene til å forutsi potensielle effekter av SNP og av mange forbindelser på CYP3A4-mediert metabolisme (Lewis et al., 1998). For eksempel bidrar ikke-kovalente, hydrofobe, elektrostatiske og van der Waals-interaksjoner til orienteringen av en forbindelse og dermed til binding og reaksjon på et enzyms aktive sted. I sin tur vil disse bestemme enzymets affinitet og spesifisitet til forskjellige substrater, og styrken til enzymhemmere (Kirchmair et al., 2012; Basheer et al., 2017).
Her foreslår vi en ny tilnærming til å måle allelfrekvensen til CYP3A4-mutasjoner i forskjellige etniske grupper. Denne omfattende tilnærmingen har makten til å fremheve mutasjoner som er utbredt i bestemte etniske grupper, og kombinert med screening for samvirkende kjemikalier, f.eks. Hemmere fra mat, vil gjøre det mulig å belyse effekten av spesielle mutasjoner på stoff-mat-interaksjon, som fungerer som en innledende gå mot personlig medisin og ernæring. Dette arbeidet kan øke bevisstheten om den mulige kliniske betydningen av proteinendrende CYP3A4 SNP, og foreslår også noen få nødvendige verktøy for markedsføring og anvendelse av presisjon og personlig medisin.
Materialer og metoder
Databasescreening og dataanalyse
CYP3A4-variasettet ble lastet ned fra gnomAD-nettleseren2 som en CVS-fil. Python 2.7 med NumPy, pandaer og matplotlib-pakker ble brukt til dataanalyse og visualisering (se Supplerende datablad S1). Agglomerativ hierarkisk klynging ble utført ved hjelp av Expander 7-programvaren (Shamir et al., 2005) med Pearson rang-korrelasjonskoeffisienten som et mål på likheter og fullstendig koblingstype. En avstandsterskel på 0,6 ble satt for gruppering av SNP.
I silico ble Polymorphism Modelling
Maestro 2017-2 release (Schrodinger, New York, NY, USA) brukt til beregningsmodellering. CYP3A4-dockingmodell ble bygget som tidligere beskrevet (Basheer et al., 2017). Kort fortalt ble CYP3A4-krystallstruktur (PDB-oppføring 2V0M) behandlet, modifisert og raffinert etter trinnene i veiviseren for proteinberedning. Et docking-gitter med en metallkoordinasjonsbegrensning for Fe2 + i hemgruppen ble generert basert på sentroid av ketokonazol i det originale bindingsstedet i krystallstrukturen. Syv mutasjoner ble valgt for docking-simuleringer, en som representant for hver etnisk gruppe (tabell 1, 2). For hvert variantprotein ble en enkeltpunktsmutasjon introdusert før trinnene for proteinfremstilling. 3D strukturer av ligander ble generert basert på 2D strukturer fra PubChem3 og forberedt for docking ved hjelp av LigPrep oppgave.OPLS3 kraftfelt og standard Glide-alternativer for standard presisjon ble brukt for dockingmodellen, med unntak av at metallkoordinasjonsbegrensningen ble brukt, samt 30 poser for antall poser som skal inkluderes og 10 poser for antall poser å skrive ute. For hver ligand ble dockingsresultatet med den laveste Glide-emodellpoeng valgt.

Tabell 1. Valgte representative SNP for syv etniske grupper.

Tabell 2. Frekvens (%) av utvalgte mutasjoner etter etnisk gruppe.
Resultater
Genome Aggregation Database (gnomAD; se tekstfotnote 2) samler både exome- og genom-sekvenseringsdata fra et bredt utvalg av storskala sekvenseringsprosjekter. Den inkluderer data fra 125.748 eksomsekvenser og 15.708 helgenomsekvenser fra 141.456 ikke-relaterte individer som representerer syv etniske populasjoner (Lek et al., 2016). GnomAD-databasen presenterer 856 varianter av CYP3A4, hvorav 397 er introniske og så mange som 459 er eksoniske. Av de eksoniske SNP-ene er 312 missense-mutasjoner, noe som indikerer at de påvirker proteinstrukturen. CYP3A4-genet er 34 205 bp langt. De 13 eksonene består av en kodingsregion på 1,512 bp som produserer et protein med 504 aminosyrer. De 412 eksoniske SNPene med unike posisjoner i dette genet resulterer i en eksonisk SNP-tetthet på 272 / kbp (tilleggstabell S1).
Beregning av differensialallelfrekvenser per etnisk gruppe avslører at noen populasjoner viser høyere frekvenser av mutasjoner (Figur 1A). De fleste av CYP3A4-mutasjonene i den europeiske befolkningen er virkelig sjeldne, som man vanligvis tror, mens mutasjoner i andre populasjoner, som afrikansk og østasiatiske, er mye mer utbredt (tilleggstabell S2).
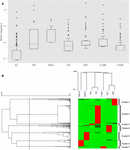
Figur 1. Analyse av CYP3A4 missense SNPs i syv forskjellige populasjoner. (A) Log-skala boksplott av allelfrekvenser. Ruter representerer interkvartilområdet (IQR), blå linjer representerer medianene, kinnskjegg representerer data innenfor 1,5 IQR og avvik er vist som små sirkler. (B) Hierarkisk gruppering av allelfrekvenser. Hver rad representerer en enkelt SNP. Hver kolonne representerer distinkt etnisk befolkning. Allelfrekvensen til SNPene i hver av populasjonene er representert av fargen på den tilsvarende cellen i matrisefilen. Grønt og rødt representerer henholdsvis lav og høy frekvens. Det øvre dendrogrammet viser likheter i allelfrekvensmønsteret mellom hver gruppe fag. Venstre dendrogram representerer gruppering av gener i to grupper. Den stiplede linjen representerer 0,6 avstandsterskelen som brukes til å dele seg til grupper. EU – europeisk (ikke-finsk; n = 64 603), FIN – europeisk (finsk; n = 12 562), ASH J – Ashkenazi-jødisk (n = 5 185), LTN – latino (n = 17 720), AFR – afrikansk (n = 12,487), E ASN – østasiatiske (n = 9 977), S ASN – sørasiatiske (n = 64 603).
Vi brukte hierarkisk gruppering for å gruppere varianter med lignende frekvensmønstre. Vår dataanalyse ga syv forskjellige klynger (figur 1B). Videre observeres det tydelig at høyfrekvente SNP i hver klynge er karakteristiske for en spesifikk populasjon. Hierarkisk klyngeanalyse av de etniske gruppene støtter sammenhengen mellom genetisk varians og etnisitet ved å gruppere relaterte etnisiteter som sør- og østasiatere samt finske og ikke-finske europeere.
En beregningsmodell ble brukt til å vurdere mulig påvirkning av punktmutasjoner i CYP3A4 på dens evne til å binde substrater og hemmere. CYP3A4 er i stand til å oksidere et bredt spekter av endogene og xenobiotiske forbindelser. Her ble ketokonazol valgt som et representativt medikament og en meget effektiv spesifikk hemmer; androstenedion og testosteron ble valgt som representativt endogent hormon; og demetoksykurcumin og epigallocatechin ble valgt som representanter for diettbioaktive stoffer. En dockingsmodell ble bygget for å forutsi bindingsposisjonene til de valgte forbindelsene i CYP3A4-bindingsstedet. Modellen ble først validert ved vellykket å gjenopprette ketokonazolposen i bindingsstedet, med en RMSD på 1,52 Å i forhold til den opprinnelige krystallstrukturen. Syv mutante proteiner ble designet basert på krystallstrukturen til villtypeproteinet (tilleggs figur S1). For hver etnisk gruppe ble den hyppigste unike mutasjonen valgt som representativ. Effekten av enkeltmutasjoner på substratbindingen ble vurdert basert på sammenligningen mellom forankringsposer på det opprinnelige proteinet og på variantproteiner. Endringer i dockingposisjoner når det gjelder RMSD er oppsummert i tabell 3.

Tabell 3. RMSD i forhold til WT for dockingligander til bindingssteder av syv CYP3A4-varianter.
Effekten av CYP3A4 SNP på substratbinding ble funnet å være mutasjons-substratspesifikk. Bare i noen få tilfeller forårsaket mutasjoner en endring i bindingsposisjonen til en ligand i bindingslommen. Testosteron docking positur var den samme i alle syv testede varianter. E262K-, D174H- og K168N-varianter forårsaket ikke en bindende positurendring i noen av de testede molekylene. L373F- og T163A-mutasjonene endret imidlertid bindingsposisjonen til androstenedion slik at den ble plassert parallelt med hemgruppen i stedet for vinkelrett på den, som i WT-proteinet. Androstenedion ble også rotert slik at cyklopentanon-gruppen er lokalisert proksimalt til hem, i stedet for cykloheksanon-gruppen i WT-proteinet. S222P- og L293P-mutasjonene forårsaket bare en liten rotasjon i bindingsposisjonen til androstenedion (figur 2A). Av alle undersøkte mutasjoner forårsaket bare S222P vesentlige endringer i forankringsposene til ketokonazol og demetoksykurkumin på bindingsstedet (figur 2B, C); mens for epigallocatechin var den positurendrende mutasjonen L373F (figur 2D).
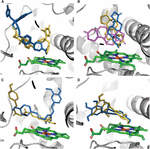
Figur 2. Modeller av ligander forankret ved bindingsstedet til CYP3A4. (A) Ketokonazol, (B) androstenedion, (C) demetoksykurkumin og (D) epigallokatekin. Proteinbindingsstedet er representert med grå bånd; heme er representert av grønne pinner, forankringsposer i WT-proteinet og i S222P og L373F er mutanter vist som henholdsvis oransje, blå og fiolett pinner. Androstenedione-dockingposer i L293P og i T136A-varianter overlapper poseringene i henholdsvis S222P og i L373F-varianter.
Diskusjon
Cytokrom P450 3A4 er det viktigste enzymet som er ansvarlig for mat-legemiddelinteraksjoner. Nåværende forskning på mutasjoner i CYP3A4 har vært fokusert på noen få dusin SNPer funnet i utpekte studier (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. ., 2002; Murayama et al., 2002). Som demonstrert her representerer de toppen av et isfjell med tanke på utbredelsen og potensielle utfall av CYP3A4-mutasjoner. Overfloden av store genom- og exome-sekvenseringsprosjekter har åpnet en ny vei for identifisering av mange ukjente mutasjoner. Her viser vi at de tidligere presenterte mutasjonene bare er toppen av isfjellet, ved å demonstrere 856 mutasjoner som finnes i CYP3A4, hvorav en tredjedel endrer proteinstrukturen. Ved hjelp av en kohort på 141 456 ikke-relaterte individer ble nøyaktige allelfrekvenser av CYP3A4-mutasjoner beregnet for syv separate etnisiteter. Så langt vi kjenner til, er dette den største og mest omfattende datastudien av CYP3A4-eksoniske mutasjoner og deres allelfrekvenser i forskjellige populasjoner, publisert til dags dato.
Polymorfe CYP3A4-enzymer kan være veldig viktige for å forklare forskjeller i legemiddeleffektivitet og toksisitet blant forskjellige individer. Mutasjoner i CYP3A4-genet kan føre til avskaffet, redusert, endret eller økt enzymatisk aktivitet. Eksoniske mutasjoner kan endre enzymatisk aktivitet, som det er vist i noen få kliniske studier med utvalgte substrater. Noen tilfeller av endret metabolisme på grunn av SNP i CYP3A4 er allerede beskrevet i litteraturen (Eiselt et al., 2001; Miyazaki et al., 2008). Til tross for SNPs funksjonelle betydning og kliniske relevans i CYP3A4 og muligens på grunn av deres relativt lave identifiserte frekvens i den generelle befolkningen, har polymorfisme i CYP3A4 ikke fått den oppmerksomheten den fortjener.
Her tjente syv mutasjoner til å forutsi effekten av SNPs på substrat- og hemmerbindingsorientering. I litteraturen deler CYP3A4 polymorfisme den generelle befolkningen i tre grupper – dårlige metaboliserere, normale metaboliserere og raske metabolisatorer, basert på introniske SNPer som endrer ekspressjonsnivåer i stedet for struktur (Zanger og Schwab, 2013). Våre beregninger antyder en tilleggsklassifisering: de endrede metabolisatorene. Noen mutasjoner foreslått av vår virtuelle modell vil føre til en endring i bindingsorienteringen til individuelle ligander. Disse endringene forventes å redusere sannsynligheten for enzymatisk oksidasjon på grunn av økt avstand fra hem, eller føre til produkter som ellers ikke vil være tydelige under toksisitetstester utført som en del av medisinutviklingsprosessen. Imidlertid, som vår modell forutsier, er CYP3A4-mutasjoner godartede for de fleste substrater.
Modifisert posisjon av et substrat i bindingslommen på grunn av proteinstrukturell endring er bare en mulig mekanisme der en mutasjon kan endre et proteins aktivitet . Nedsatt forankring av proteinet til membranen, ødelagte substratledende kanaler og kompromittert utgang av produktene gir ytterligere mekanismer for en mutasjonsendring i proteinets aktivitet. Som vist her er effekten av hver mutasjon substratspesifikk.Å bestemme hvilke kombinasjoner av substrater og mutasjoner som kan endre den enzymatiske aktiviteten ved å bruke tradisjonelle in vitro-metoder er arbeidskrevende, og understreker behovet for prediktive virtuelle verktøy for å løse dette komplekse puslespillet.
Offentlig og profesjonell interesse for personlig medisin og presisjonsmedisin vokser raskt. Prediksjon av modifisert stoffskifte basert på individuell polymorfisme i CYP3A4 ser ut til å være bare et spørsmål om tid. Her foreslår vi at forskjellige etniske grupper bærer unike sett med CYP3A4 SNP. Faktisk kan etnisitet tjene som et første gjennomførbare trinn i personlig medisin, før implementeringen av et individuelt DNA-skjermbilde for alle. Interessant, etnisitet har ytterligere en implikasjon for CYP3A4 stoffskifte, og er en viktig faktor i å bestemme matvalg og kostvaner. Det kan foreslås at terapeutiske regimer skal utformes spesielt for hver etnisk gruppe, i det minste for legemidler som er sterkt metabolisert av CYP3A4. Dette fremhever mulighetene for å utnytte og integrere databaser og dyp læring for å identifisere hvordan SNP, etnisitet, diettforbindelser og medisiner endrer CYP3A4-aktiviteten og suksessen til et medisinsk regime.
Datatilgjengelighet
Offentlig tilgjengelige datasett ble analysert i denne studien. Disse dataene finner du her: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Forfatterbidrag
Alle oppførte forfattere har gitt et betydelig, direkte og intellektuelt bidrag til arbeid, og godkjente det for publisering.
Erklæring om interessekonflikt
Forfatterne erklærer at forskningen ble utført i fravær av kommersielle eller økonomiske forhold som kunne tolkes som potensielle. interessekonflikt.
Supplerende materiale
FIGUR S1 | 3D-båndmodell av CYP3A4 og plasseringen av de muterte aminosyrene i de syv variantproteinene designet for docking. Heme er representert som grønne pinner, Fe2 + er representert som en rød sfære, SNPs brukt i in silico-analyse er representert som røde områder på båndet og R-grupper av muterte aminosyrer i variantmodeller vises eksplisitt som lysegrå pinner.
TABELL S1 | CYP3A4 SNP-typer i en befolkning på 141, 456 ikke-relaterte individer som representerer 7 etniske populasjoner.
TABELL S2 | CYP3A4 SNPs etter etnisk gruppe.
Fotnoter
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov