Inledning
Under årtionden har interaktioner mellan mat och läkemedel (FDI) och växter-läkemedelsinteraktioner varit kända för att begränsa framgången för medicinska behandlingar. Det enorma antalet möjliga interaktioner mellan genetiska variationer, medicinska regimer och de många bioaktiva föreningarna som finns i mat och örter resulterar i överväldigande komplexitet. Moderna verktyg som stor-dataanalys, maskininlärning och simulering av protein-ligand-interaktioner kan hjälpa oss att svara på en hel uppsättning frågor: Kan matval bidra till att terapeutiska regimer misslyckas och i så fall hur? Vilken eller vilka livsmedel bör konsumeras innan du tar ett förskrivet läkemedel? Och förmodligen den mest spännande frågan: Hur kan vi använda dessa verktyg för att förutsäga personlig FDI? Det är uppenbart att många svar ligger i metabolismen av läkemedel, livsmedel och örter med cytokrom P450 3A4 (CYP3A4) i levern och mag-tarmkanalen (Galetin et al., 2010; Basheer och Kerem, 2015).
Majoriteten av gener som kodar för CYP-enzymer är polymorfa. Hittills är den mest omfattande informationskällan som beskriver CYP-alleler Pharmacogen Variation Consortium1, där färre än 100 alleler av CYP3A4 är representerade. Av dessa är färre än 40 exoniska SNP (enkla nukleotidpolymorfismer) som resulterar i en modifierad proteinsekvens. Det lilla antalet försökspersoner i alla tidigare publicerade arbeten med CYP3A4-mutationer ger oss begränsad data om verkliga frekvenser av CYP3A4-mutationer i hela befolkningen och i definierade grupper. , dess kliniska konsekvenser är fortfarande oklara i de flesta fall (Zanger et al., 2014). Att förstå vilka och när SNP kan ha klinisk betydelse är en oerhört komplex uppgift. In vitro-analyser är tidskrävande, dyra och praktiskt taget av låg relevans med tanke på den stora mängden mutationer och det oändliga antalet mat-läkemedelskombinationer. Molekylära modelleringsmetoder, inklusive docknings- och fria energibindningsberäkningar, kan tjäna till att förutsäga potentiella effekter av SNP och av många föreningar på CYP3A4-medierad metabolism (Lewis et al., 1998). Till exempel bidrar icke-kovalenta, hydrofoba, elektrostatiska och van der Waals-interaktioner till orienteringen av en förening och därmed till dess bindning och reaktion vid ett enzyms aktiva plats. I sin tur kommer dessa att avgöra enzymets affinitet och specificitet för olika substrat och styrkan hos enzymhämmare (Kirchmair et al., 2012; Basheer et al., 2017).
Här föreslår vi en ny metod för att mäta allelfrekvensen för CYP3A4-mutationer i olika etniska grupper. Detta omfattande tillvägagångssätt har förmågan att lyfta fram mutationer som förekommer i speciella etniska grupper, och i kombination med screening för interagerande kemikalier, t.ex. hämmare från mat, gör det möjligt att klargöra effekterna av vissa mutationer på läkemedelsinteraktion, som fungerar som en initial steg mot personlig medicin och näring. Detta arbete kan öka medvetenheten om den möjliga kliniska betydelsen av proteinförändrande CYP3A4 SNP och föreslår också några nödvändiga verktyg för att främja och tillämpa precision och personlig medicin.
Material och metoder
Databasscreening och dataanalys
CYP3A4-varianteruppsättningen laddades ner från gnomAD-webbläsaren2 som en CVS-fil. Python 2.7 med NumPy, pandor och matplotlib-paket användes för dataanalys och visualisering (se Kompletterande datablad S1). Agglomerativ hierarkisk klustering utfördes med hjälp av mjukvaran Expander 7 (Shamir et al., 2005) med Pearson rank korrelationskoefficient som ett mått på likheter och fullständig kopplingstyp. Avståndströskeln på 0,6 sattes för gruppering av SNP.
I silico Polymorphism Modelling
Maestro 2017-2 release (Schrodinger, New York, NY, USA) användes för beräkningsmodellering. CYP3A4-dockningsmodellen byggdes som beskrivits tidigare (Basheer et al., 2017). I korthet behandlades, modifierades och raffinerades CYP3A4-kristallstruktur (PDB-post 2V0M) enligt steget för proteinberedningsguiden. Ett dockningsgaller med en metallkoordinationsbegränsning för Fe2 + i hemgruppen genererades baserat på ketokonazols centroid i det ursprungliga bindningsstället i kristallstrukturen. Sju mutationer valdes för dockningssimuleringar, en som en representant för varje etnisk grupp (Tabell 1, 2). För varje variantprotein infördes en enda punktsmutation före proteinberedningsstegen. 3D-strukturer av ligander genererades baserat på 2D-strukturer från PubChem3 och förbereddes för dockning med LigPrep-uppgift.OPLS3-kraftfält och standardglidalternativ för standardprecision tillämpades för dockningsmodellen, med undantaget att metallkoordinationsbegränsningen användes, liksom 30 poser för antalet poser som ska inkluderas och 10 poser för antalet poser att skriva ut. För varje ligand valdes dockningsresultatet med lägsta Glide-emodelpoäng.

Tabell 1. Valda representativa SNP för sju etniska grupper.

Tabell 2. Frekvens (%) av valda mutationer efter etnisk grupp.
Resultat
Genomens aggregeringsdatabas (gnomAD; se fotnot 2) samlar både exome- och genom-sekvenseringsdata från en mängd olika storskaliga sekvenseringsprojekt. Den innehåller data från 125.748 exomsekvenser och 15.708 helgenomsekvenser från 141.456 orelaterade individer som representerar sju etniska populationer (Lek et al., 2016). GnomAD-databasen presenterar 856 varianter av CYP3A4, varav 397 är introniska och så många som 459 exoniska. Av de exoniska SNP: erna är 312 missensmutationer, vilket indikerar att de påverkar proteinstrukturen. CYP3A4-genen är 34205 bp lång. Dess 13 exoner omfattar en kodande region på 1 512 bp som producerar ett protein med 504 aminosyror. De 412 exoniska SNP: erna med unika positioner i denna gen resulterar i en exonisk SNP-densitet på 272 / kbp (kompletterande tabell S1).
Beräkning av differentiella allelfrekvenser per etnisk grupp visar att vissa populationer uppvisar högre frekvenser av mutationer (Figur 1A). De flesta av CYP3A4-mutationerna i den europeiska befolkningen är verkligen sällsynta, vilket man ofta tror, medan mutationer i andra populationer, såsom afrikanska och östasiatiska, är mycket vanligare (kompletterande tabell S2).
div>
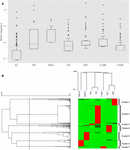
Figur 1. Analys av CYP3A4 missense SNP i sju olika populationer. (A) Log-skala rutdiagram över allelfrekvenser. Rutor representerar interkvartilintervallet (IQR), blå linjer representerar medianerna, morrhår representerar data inom 1,5 IQR och outliers visas som små cirklar. (B) Hierarkisk gruppering av allelfrekvenser. Varje rad representerar en enda SNP. Varje kolumn representerar distinkt etnisk befolkning. Allelfrekvensen för SNP: erna i var och en av populationerna representeras av färgen på motsvarande cell i matrisfilen. Grönt och rött representerar låg respektive hög frekvens. Det övre dendrogrammet visar likheter i allelfrekvensmönstret mellan varje grupp av försökspersoner. Det vänstra dendrogrammet representerar gruppering av gener i två grupper. Den streckade linjen representerar 0,6 avståndströskeln som används för delning till grupper. EU – europeisk (icke-finsk; n = 64 603), FIN – europeisk (finsk; n = 12 562), ASH J – Ashkenazi judisk (n = 5 185), LTN – latino (n = 17 720), AFR – afrikansk (n = 12,487), E ASN – östasiatiska (n = 9 977), S ASN – sydasiatiska (n = 64 603).
Vi använde hierarkisk gruppering för att gruppera varianter med liknande frekvensmönster. Vår dataanalys gav sju olika kluster (figur 1B). Vidare observeras det tydligt att högfrekventa SNP i varje kluster är karakteristiska för en specifik population. Hierarkisk klusteranalys av de etniska grupperna stöder sambandet mellan genetisk varians och etnicitet genom att gruppera relaterade etniciteter som södra och östasiatiska samt finska och icke-finska européer.
En beräkningsmodell användes för att bedöma det möjliga inflytandet av punktmutationer i CYP3A4 på dess förmåga att binda substrat och hämmare. CYP3A4 kan oxidera ett brett spektrum av endogena och xenobiotiska föreningar. Här valdes ketokonazol som ett representativt läkemedel och en mycket effektiv specifik hämmare; androstenedion och testosteron valdes som representativt endogent hormon; och demetoxikurcumin och epigallocatechin valdes som representanter för dietbioaktiva ämnen. En dockningsmodell byggdes för att förutsäga bindningsställen för de valda föreningarna i CYP3A4-bindningsstället. Modellen validerades först genom att framgångsrikt återställa ketokonazolpositionen i bindningsstället, med en RMSD på 1,52 Å relativt den ursprungliga kristallstrukturen. Sju mutanta proteiner designades baserat på kristallstrukturen hos vildtypsproteinet (kompletterande figur S1). För varje etnisk grupp valdes den vanligaste unika mutationen som representativ. Effekten av enstaka mutationer på substratbindningen bedömdes baserat på jämförelsen mellan dockningsposer på det nativa proteinet och på variantproteiner. Ändringar i dockningsställningar i termer av RMSD sammanfattas i tabell 3.

Tabell 3. RMSD relativt WT för dockningsligander till bindningsställen för sju CYP3A4-varianter.
Effekten av CYP3A4 SNP på substratbindning befanns vara mutationssubstratspecifik. Endast i ett fåtal fall orsakade mutationer en förändring i bindningsposen hos en ligand i bindningsfickan. Testosteron dockning pose var densamma i alla sju testade varianter. E262K-, D174H- och K168N-varianterna orsakade inte en bindningsförändring i någon av de testade molekylerna. Emellertid ändrade L373F- och T163A-mutationerna bindningsposen för androstenedion så att den placerades parallellt med hemgruppen snarare än vinkelrätt mot den, som i WT-proteinet. Androstenedion roterades också så att cyklopentanon-gruppen är belägen proximalt till hem istället för cyklohexanon-gruppen i WT-proteinet. S222P- och L293P-mutationerna orsakade endast en liten rotation i androstenedionens bindande ställning (figur 2A). Av alla undersökta mutationer orsakade endast S222P väsentliga förändringar i dockningsställen för ketokonazol och demetoxikurkumin vid bindningsstället (figur 2B, C); medan för epigallocatechin var den positivt förändrade mutationen L373F (figur 2D).
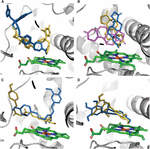
Figur 2. Modeller av ligander dockade vid bindningsstället för CYP3A4. (A) Ketokonazol, (B) androstenedion, (C) demetoxikurcumin och (D) epigallocatechin. Proteinbindningsstället representeras av grå band; heme representeras av gröna pinnar, dockningsställningar i WT-proteinet och i S222P- och L373F-mutanter visas som orange, blå respektive violetta pinnar. Androstenedione-dockningsposer i L293P och i T136A-varianter överlappar poserna i S222P respektive i L373F-varianter.
Diskussion
Cytokrom P450 3A4 är det huvudsakliga enzymet som ansvarar för mat-läkemedelsinteraktioner. Aktuell forskning om mutationer i CYP3A4 har fokuserats på några dussin SNP som hittats i utsedda studier (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. ., 2002; Murayama et al., 2002). Som visas här representerar de toppen av ett isberg med tanke på prevalensen och potentiella resultat av CYP3A4-mutationer. Överflödet av stora genom- och exomsekvenseringsprojekt har öppnat en ny väg för identifiering av många okända mutationer. Här visar vi att de tidigare presenterade mutationerna bara är toppen av isberget genom att visa 856 mutationer som finns i CYP3A4, varav en tredjedel modifierar proteinstrukturen. Med hjälp av en kohort av 141 456 oberoende individer beräknades exakta allelfrekvenser för CYP3A4-mutationer för sju separata etniciteter. Så vitt vi vet är detta den största och mest omfattande stora datastudien av CYP3A4 exoniska mutationer och deras allelfrekvenser i olika populationer, publicerade hittills.
Polymorfa CYP3A4-enzymer kan vara mycket viktiga för att förklara skillnader i läkemedelseffektivitet och toxicitet hos olika individer. Mutationer i CYP3A4-genen kan leda till avskaffad, minskad, förändrad eller ökad enzymatisk aktivitet. Exoniska mutationer kan modifiera enzymatisk aktivitet, vilket har visats i några kliniska studier med utvalda substrat. Vissa fall av förändrad metabolism på grund av SNP i CYP3A4 har redan beskrivits i litteraturen (Eiselt et al., 2001; Miyazaki et al., 2008). Trots SNPs funktionella betydelse och kliniska relevans i CYP3A4 och möjligen på grund av deras relativt låga identifierade frekvens i allmänheten har polymorfism i CYP3A4 inte fått den uppmärksamhet som den förtjänar.
Här har sju mutationer tjänat till att förutsäga effekten av SNP på substrat- och inhibitorbindningsorientering. I litteraturen delar CYP3A4 polymorfism den allmänna befolkningen i tre grupper – dåliga metaboliserare, normala metaboliserare och snabba metaboliserare, baserat på introniska SNP som modifierar uttrycksnivåer snarare än struktur (Zanger och Schwab, 2013). Våra beräkningar föreslår en ytterligare klassificering: de förändrade metaboliserarna. Vissa mutationer som föreslås av vår virtuella modell skulle orsaka en förändring i bindningsorienteringen för enskilda ligander. Dessa förändringar kan förväntas minska sannolikheten för enzymatisk oxidation på grund av ökat avstånd från hem, eller leda till produkter som annars inte skulle vara uppenbara under toxicitetstester som utförts som en del av läkemedelsutvecklingsprocessen. Men som vår modell förutspår, är CYP3A4-mutationer för de flesta substrat godartade.
Modifierat läge för ett substrat i bindningsfickan på grund av proteinstrukturell förändring är bara en möjlig mekanism genom vilken en mutation kan förändra proteinets aktivitet . Nedsatt förankring av proteinet till membranet, skadade substratledande kanaler och komprometterat utträde av produkterna ger ytterligare mekanismer för en mutationsförändring i proteinets aktivitet. Som visas här är effekten av varje mutation substratspecifik.Att bestämma vilka kombinationer av substrat och mutationer som kan modifiera den enzymatiska aktiviteten med traditionella in vitro-metoder är mödosamt och betonar behovet av prediktiva virtuella verktyg för att lösa detta komplexa pussel.
Allmänt och professionellt intresse för personlig medicin och precisionsmedicin växer snabbt. Förutsägelse av modifierad läkemedelsmetabolism baserat på individuell polymorfism i CYP3A4 verkar bara vara en tidsfråga. Här föreslår vi att distinkta etniska grupper bär unika uppsättningar av CYP3A4 SNP. Faktum är att etnicitet kan fungera som ett första genomförbart steg i personlig medicin, före implementeringen av en individuell DNA-skärm för alla. Intressant nog har etnicitet ytterligare en implikation för CYP3A4-läkemedelsmetabolism, vilket är en viktig faktor för att bestämma livsmedelsval och kostvanor. Det kan föreslås att terapeutiska regimer ska utformas specifikt för varje etnisk grupp, åtminstone för läkemedel som starkt metaboliseras av CYP3A4. Detta belyser möjligheterna att utnyttja och integrera databaser och djupinlärning för att identifiera hur SNP, etnicitet, dietföreningar och läkemedel modifierar CYP3A4-aktivitet och framgången för ett medicinskt system.
Datatillgänglighet
Allmänt tillgängliga datamängder analyserades i denna studie. Dessa uppgifter kan hittas här: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Författarens bidrag
Alla listade författare har gjort ett betydande, direkt och intellektuellt bidrag till arbetet och godkände det för publicering.
Uttalande av intressekonflikter
Författarna förklarar att forskningen genomfördes i avsaknad av kommersiella eller ekonomiska relationer som skulle kunna tolkas som en potential intressekonflikt.
Kompletterande material
FIGUR S1 | 3D-bandmodell av CYP3A4 och placeringen av de muterade aminosyrorna i de sju variantproteinerna utformade för dockning. Heme representeras som gröna pinnar, Fe2 + representeras som en röd sfär, SNP som används i silico-analysen representeras som röda områden på bandet och R-grupper av muterade aminosyror i variantmodeller visas uttryckligen som ljusgrå pinnar.
TABELL S1 | CYP3A4 SNP-typer i en befolkning på 141, 456 oberoende individer som representerar 7 etniska befolkningar.
TABELL S2 | CYP3A4 SNP efter etnisk grupp.
Fotnoter
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov