Denna teknik utvecklades i slutet av 1980-talet och är en kraftfull metod för att detektera translokationer (omarrangemang bland kromosomer).
För utvecklingen av FISH var det nödvändigt att isolera varje mänsklig kromosom. Därefter fragmenterades DNA från dessa kromosomer och placerades i bakterieceller för att förstärka det (producera många kopior). På detta sätt kan ett stort antal kopior av DNA från varje kromosom erhållas.
Dessa amplifierade DNA-fragment är märkta med lämpliga fluorescerande (ljusemitterande) färgämnen och får hybridisera (fästa) till metafaskromosomer . De fluorescerande märkta DNA: n kommer att fästa vid de analoga kromosomerna från vilka de härstammar. (DNA-fragment med samma bassekvenser har den egenskapen att de fäster vid varandra.)
På detta sätt, om en del av en målad kromosom (till exempel gul) hade genomgått ett utbyte med en annan, icke -målade kromosomer (färgade röda) är det möjligt att detektera aberrationen (kallas en ömsesidig translokation) eftersom den avvikande kromosomen innehåller både gula och röda segment. Vanligtvis kan ett par tvåfärgade kromosomer detekteras i ett metafas eftersom två kromosomer vanligtvis byter ut en del av deras DNA. material, till exempel med Giemsa. Tänk dig till exempel ett fall där de två utbytta segmenten hade samma längder. De två translokerade kromosomerna ska se ut som helt normala, både i form och längd. Om vi använder FISH kan dock sådan translokation tydligt upptäckas.
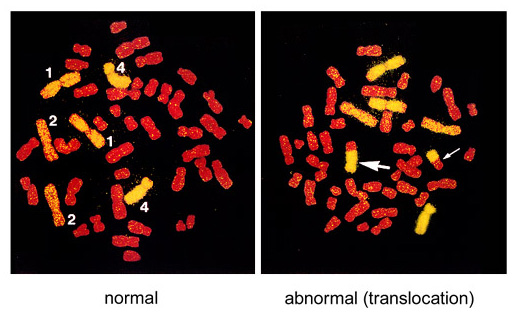
Figur. Ett exempel på FISH-behandlade metafaskromosomer
Här märktes kromosomer 1, 2 och 4 gula med FISH och de andra kromosomerna färgades rött. Translokationer mellan gula och röda kromosomer detekteras. Den vänstra bilden representerar en normal cell (siffrorna i figuren indikerar kromosomnummer) och den högra bilden är ett exempel på ömsesidig translokation med två tvåfärgade kromosomer (indikeras av två pilar).