Bevezetés
Évtizedek óta ismert, hogy az élelmiszer-gyógyszer kölcsönhatások (FDI) és a gyógynövények közötti kölcsönhatások korlátozzák az orvosi kezelések sikerét. A genetikai variációk, az orvosi rezsimek, valamint az élelmiszerekben és a gyógynövényekben található számos bioaktív vegyület közötti rendkívül sok lehetséges kölcsönhatás elsöprő bonyolultságot eredményez. A modern eszközök, mint például a nagy adatok elemzése, a gépi tanulás és a fehérje-ligandum kölcsönhatások szimulációja, segítséget nyújthatnak számunkra a kérdések egész sorának megválaszolásában: Az ételválasztás hozzájárulhat a terápiás rendszerek kudarcához, és ha igen, hogyan? Mely ételeket kell fogyasztani az előírt gyógyszer bevétele előtt? És valószínűleg a legizgalmasabb kérdés: Hogyan használhatjuk ezeket az eszközöket a személyes FDI előrejelzésére? Nyilvánvaló, hogy sok válasz abban rejlik, hogy a májban és az emésztőrendszerben a citokróm P450 3A4 (CYP3A4) metabolizálja a gyógyszereket, ételeket és gyógynövényeket (Galetin et al., 2010; Basheer és Kerem, 2015).
A CYP enzimeket kódoló gének többsége polimorf. Napjainkig a legátfogóbb információforrás, amely részletezi a CYP allélokat, a Pharmacogene Variation Consortium1, amelyben kevesebb, mint 100 CYP3A4 allél van jelen. Ezek közül kevesebb, mint 40 exonikus SNP (egy nukleotid polimorfizmus), amelyek módosított fehérjeszekvenciát eredményeznek. A CYP3A4 mutációkról szóló, korábban publikált összes munka alanyainak száma korlátozott, és korlátozott adatokkal szolgál a CYP3A4 mutációk valódi gyakoriságáról az egész populációban és meghatározott csoportokban.
Nemcsak az SNP-k előfordulásáról szóló megbízható információk hiányosak , klinikai vonatkozásaik a legtöbb esetben még nem tisztázottak (Zanger et al., 2014). Rendkívül összetett feladat annak megértése, hogy mely SNP-knek és mikor lehet klinikai jelentőségük. Az in vitro vizsgálatok időigényesek, drágák és gyakorlatilag alacsony jelentőségűek, figyelembe véve a mutációk nagy mennyiségét és az élelmiszer-gyógyszer kombinációk végtelen számát. A molekuláris modellezési módszerek, beleértve a dokkolást és a szabadenergia-megkötési számításokat is, megjósolhatják az SNP-k és számos vegyület potenciális hatásait a CYP3A4 által közvetített metabolizmusra (Lewis et al., 1998). Például a nem kovalens, hidrofób, elektrosztatikus és van der Waals kölcsönhatások mind hozzájárulnak a vegyület orientációjához, és ezáltal annak kötéséhez és reakciójához egy enzim aktív helyén. Ezek viszont meghatározzák az enzim affinitását és specificitását a különböző szubsztrátokhoz, valamint az enzim inhibitorok hatékonyságát (Kirchmair et al., 2012; Basheer et al., 2017).
Itt egy új javaslatot javasolunk. megközelítés a különböző etnikai csoportokban a CYP3A4 mutációk allél gyakoriságának mérésére. Ez az átfogó megközelítés arra képes, hogy kiemelje az adott etnikai csoportokban elterjedt mutációkat, és kölcsönhatásba lépő vegyi anyagok, például az ételből származó inhibitorok szűrésével kombinálva lehetővé teszi az egyes mutációk gyógyszer-étel kölcsönhatásra gyakorolt hatásainak tisztázását, amely kiindulópontként szolgál lépés a személyre szabott orvoslás és táplálkozás felé. Ez a munka felhívhatja a figyelmet a fehérjét megváltoztató CYP3A4 SNP-k lehetséges klinikai jelentőségére, és néhány szükséges eszközt is javasol a precíziós és személyre szabott orvoslás népszerűsítéséhez és alkalmazásához.
Anyagok és módszerek
Adatbázis-szűrés és adatelemzés
A CYP3A4 variánsok adatkészletét a gnomAD böngésző2 CVS fájlként töltötte le. Az adatelemzéshez és a vizualizációhoz a Python 2.7-et NumPy, pandas és matplotlib csomagokkal használták (lásd az S1 kiegészítő adatlapot). Az agglomeratív hierarchikus klaszterezést az Expander 7 szoftver (Shamir et al., 2005) alkalmazásával, a Pearson rang korrelációs együtthatóval végeztük, amely a hasonlóságok és a teljes kapcsolattípus mérésére szolgál. Az SNP-k csoportosításához 0,6 távolsági küszöböt állítottak be.
In silico Polymorphism Modeling
Maestro 2017-2 kiadást (Schrodinger, New York, NY, Egyesült Államok) használtunk a számítási modellezés. A CYP3A4 dokkoló modellt a korábban leírtak szerint építettük fel (Basheer et al., 2017). Röviden, a CYP3A4 kristályszerkezetet (PDB 2V0M bejegyzés) feldolgoztuk, módosítottuk és finomítottuk a Protein Preparation Wizard lépéseit követve. A kristályszerkezet eredeti kötődési helyén lévő ketokonazol centroidja alapján a hem csoportban lévő Fe2 + fémkoordinációs kényszerével rendelkező dokkoló rácsot hoztak létre. Hét mutációt választottak ki a dokkoló szimulációkhoz, egyet-egyet az egyes etnikai csoportok képviselőiként (1., 2. táblázat). Az egyes fehérjeváltozatokhoz egyetlen pont mutációt vezettek be a fehérje-előkészítési lépések előtt. A ligandumok 3D struktúráit a PubChem3 2D struktúrái alapján állítottuk elő, és a LigPrep feladat segítségével készítettük elő dokkolásra.Az OPLS3 erőtér és az alapértelmezett csúszás opciókat alkalmazták a szabványos pontosság érdekében a dokkoló modellnél, azzal a különbséggel, hogy a fém koordinációs kényszert alkalmazták, valamint 30 pózt a beépítendő pózok számához és 10 pózokat az írandó pózok számához ki. Minden ligandumhoz kiválasztották a legkisebb Glide emodel pontszámú dokkolási eredményt.

1. táblázat: Hét etnikai csoport kiválasztott reprezentatív SNP-je.

2. táblázat: A kiválasztott mutációk gyakorisága (%) etnikai csoportok szerint.
Eredmények
A Genom Aggregation Database (gnomAD; lásd a 2. lábjegyzetet) összesíti mind az exom, mind a genom szekvenálás adatait sokféle nagyszabású szekvenálási projektből. Hét etnikai populációt képviselő 125 748 exome-szekvencia és 15 708 teljes-genom-szekvencia adatait tartalmazza 141 456 rokon személytől (Lek et al., 2016). A GnomAD adatbázis a CYP3A4 856 változatát mutatja be, amelyek közül 397 intronikus és 459 exonikus. Az exonikus SNP-k közül 312 missense mutáció, ami azt jelzi, hogy befolyásolják a fehérje szerkezetét. A CYP3A4 gén 34 205 bp hosszú. 13 exonja egy 1512 bp kódoló régiót tartalmaz, amely 504 aminosavból álló fehérjét termel. A 412 exonikus SNP, amelynek ebben a génben egyedülálló pozíciói vannak, 272 / kbp exonikus SNP-sűrűséget eredményez (S1 kiegészítő táblázat).
Az etnikai csoportonkénti differenciális allélfrekvenciák kiszámítása során kiderül, hogy egyes populációknál magasabb a mutációk gyakorisága. (1A. Ábra). Az európai populációban a legtöbb CYP3A4-mutáció valóban ritka, mint általában gondolják, míg más populációkban, például afrikai és kelet-ázsiai mutációk sokkal gyakoribbak (S2 kiegészítő táblázat).
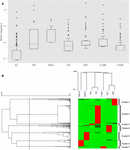
1. ábra A CYP3A4 missense SNP elemzése hét különálló populációban. (A) Az allélfrekvenciák log-skálájú dobozdiagramja. A dobozok az interkvartilis tartományt (IQR), a kék vonalak a mediánokat, a bajuszok az 1,5 IQR-en belüli adatokat mutatják, a szélső értékek pedig kis körökként jelennek meg. (B) Az allélfrekvenciák hierarchikus csoportosulása. Minden sor egyetlen SNP-t képvisel. Minden oszlop külön etnikai népességet képvisel. Az SNP-k allélfrekvenciáját az egyes populációkban a mátrixfájl megfelelő cellájának színe képviseli. A zöld és a piros alacsony, illetve magas frekvenciát jelent. A felső dendrogram hasonlóságokat mutat az allél frekvencia mintázatában az egyes alanycsoportok között. A bal dendrogram a gének csoportosulását mutatja két csoportban. A szaggatott vonal a csoportokra osztáshoz használt 0,6 távolsági küszöböt jelöli. EU – európai (nem finn; n = 64 603), FIN – európai (finn; n = 12 562), ASH J – askenázi zsidó (n = 5 185), LTN – latin (n = 17 720), AFR – afrikai (n = 12487), E ASN – kelet-ázsiai (n = 9 977), S ASN – dél-ázsiai (n = 64 603).
Hierarchikus klaszterezést használtunk hasonló frekvenciájú változatok csoportosításához. Adatelemzésünk hét különálló klasztert eredményezett (1B ábra). Továbbá egyértelműen megfigyelhető, hogy az egyes klaszterekben található magas frekvenciájú SNP-k egy adott populációra jellemzőek. Az etnikai csoportok hierarchikus klaszterelemzése alátámasztja a genetikai változatosság és az etnicitás közötti kapcsolatot azáltal, hogy csoportosítja az egymással rokon etnikumokat, például a dél- és kelet-ázsiaiakat, valamint a finn és nem finn európaiakat. a CYP3A4 pontmutációinak lehetséges hatása a szubsztrátok és inhibitorok megkötésére való képességére. A CYP3A4 képes endogén és xenobiotikus vegyületek széles skáláját oxidálni. Itt a ketokonazolt választották reprezentatív gyógyszerként és nagyon hatékony specifikus inhibitorként; az androsztenediont és a tesztoszteront reprezentatív endogén hormonként választottuk ki; az étrendi bioaktív anyagok képviselőiként pedig demetoxi-kurkumint és epigallokatechint választottak ki. Dokkoló modellt készítettek a kiválasztott vegyületek kötődési pózainak előrejelzésére a CYP3A4 kötőhelyén. A modellt először úgy erősítették meg, hogy sikeresen helyreállították a ketokonazol pózot a kötési helyen, az eredeti kristályszerkezethez viszonyítva 1,52 Å RMSD-vel. Hét mutáns fehérjét terveztek a vad típusú fehérje kristályszerkezete alapján (S1. Kiegészítő ábra). Minden etnikai csoport esetében a leggyakoribb egyedi mutációt választottuk reprezentatívnak. Az egyes mutációk hatását a szubsztrát kötődésére a dokkoló pózok natív fehérjére és variáns fehérjékre történő összehasonlítása alapján értékeltük. A dokkoló pózok változását az RMSD szempontjából a 3. táblázat foglalja össze.

3. táblázat: RMSD a ligandumok WT-jéhez viszonyítva hét CYP3A4 variáns kötési helyéhez.
A CYP3A4 SNP hatása a szubsztrátkötésre mutáció-szubsztrát specifikusnak bizonyult. Csak néhány esetben mutációk okoztak változást a ligand kötési pózában a kötő zsebben. A tesztoszteron dokkoló pózja mind a hét tesztelt változatban megegyezett. Az E262K, D174H és K168N variánsok a tesztelt molekulák egyikében sem okoztak kötési pózváltozást. Az L373F és T163A mutációk azonban megváltoztatták az androszténdion kötő pózját úgy, hogy inkább párhuzamosan helyezkedtek el a hemcsoporttal, mint merőlegesen, mint a WT fehérjében. Ezenkívül az androszténdiont úgy forgattuk, hogy a ciklopentanoncsoport a hem közelében legyen a WT fehérjében lévő ciklohexanoncsoport helyett. Az S222P és L293P mutációk csak kismértékű rotációt okoztak az androszténdion kötő pózában (2A. Ábra). Az összes vizsgált mutáció közül csak az S222P okozott lényeges változásokat a kötési helyen a ketokonazol és a demetoxi-kurkumin dokkolási helyzetében (2B, C ábra); míg az epigallokatechin esetében a pózváltó mutáció L373F volt (2D. ábra).
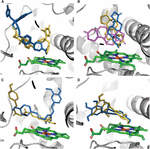
2. ábra A CYP3A4 kötődési helyén dokkolt ligandumok modelljei. (A) ketokonazol, (B) androsztendion, (C) demetoxi-kurkumin és (D) epigallokatechin. A fehérje-kötő helyet szürke szalagok képviselik; a hemet zöld pálcák képviselik, a dokkoló pózok a WT fehérjében, az S222P és L373F mutánsok pedig narancssárga, kék, illetve lila pálcikákként vannak feltüntetve. Az L293P és a T136A variánsokban található androstenedion dokkoló pózok átfedik az S222P, illetve az L373F variánsok pózait. >
A citokróm P450 3A4 az élelmiszer-gyógyszer kölcsönhatásokért felelős fő enzim. A CYP3A4 mutációinak jelenlegi kutatása néhány tucat SNP-re összpontosított, amelyeket a kijelölt tanulmányokban találtak (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. (2002; Murayama és mtsai., 2002). Amint itt bemutattuk, a jéghegy csúcsát jelentik, figyelembe véve a CYP3A4 mutációk prevalenciáját és lehetséges kimenetelét. A nagy genom és exom szekvenálás projektjeinek bősége új utat nyitott számos ismeretlen mutáció azonosítására. Itt megmutatjuk, hogy a korábban bemutatott mutációk csak a jéghegy csúcsa, bemutatva a CYP3A4-ben létező 856 mutációt, amelyek egyharmada módosítja a fehérje szerkezetét. 141 456 rokon egyénből álló kohorsz felhasználásával hét külön etnikumra kiszámolták a CYP3A4 mutációk pontos allélfrekvenciáját. Legjobb tudomásunk szerint ez a legnagyobb és legátfogóbb, a CYP3A4 exonikus mutációk és azok allél frekvenciáinak vizsgálata a különböző populációkban, a mai napig publikálva.
A polimorf CYP3A4 enzimek nagyon fontosak lehetnek a magyarázatban. különbségek a gyógyszer hatékonyságában és toxicitásában a különböző egyének között. A CYP3A4 gén mutációi megszüntethető, csökkent, megváltozott vagy fokozott enzimatikus aktivitást eredményezhetnek. Az exonikus mutációk módosíthatják az enzimatikus aktivitást, amint azt néhány klinikai vizsgálat kimutatta kiválasztott szubsztrátokkal. Az SNP-k miatt a CYP3A4-ben bekövetkező megváltozott anyagcserét néhány esetről már a szakirodalom is leírta (Eiselt et al., 2001; Miyazaki et al., 2008). Az SNP-k funkcionális fontossága és klinikai jelentősége ellenére a CYP3A4-ben, és valószínűleg az általános populációban tapasztalható viszonylag alacsony gyakoriságuk miatt, a CYP3A4 polimorfizmusa nem kapta meg a figyelmet, amelyet megérdemel.
Itt hét mutáció szolgált az előrejelzésre az SNP-k hatása a szubsztrát- és inhibitor-kötő orientációra. Az irodalomban a CYP3A4 polimorfizmus az általános populációt három csoportra osztja – gyenge metabolizálókra, normál metabolizálókra és gyors metabolizálókra, az intronikus SNP-k alapján, amelyek a szerkezet helyett az expresszió szintjét módosítják (Zanger és Schwab, 2013). Számításaink további osztályozást javasolnak: a megváltozott metabolizátorok. A virtuális modellünk által javasolt egyes mutációk változást okoznának az egyes ligandumok kötési orientációjában. Ezek a változások várhatóan csökkentik az enzimatikus oxidáció valószínűségét a hemtől való nagyobb távolság miatt, vagy olyan termékekhez vezetnek, amelyek egyébként nem lennének nyilvánvalóak a gyógyszerfejlesztési folyamat részeként végzett toxicitási vizsgálatok során. Mint azonban modellünk előrejelzi, a legtöbb szubsztrát esetében a CYP3A4 mutációk jóindulatúak.
A szubsztrát módosulási helye a kötődési zsebben a fehérje szerkezeti változásai miatt csak egy lehetséges mechanizmus, amellyel a mutáció megváltoztathatja a fehérje aktivitását . A fehérje membránhoz való horgonyzásának károsodása, a szubsztrátot vezető csatornák sérülése és a termékek kompromittált kilépése további mechanizmusokat jelent a fehérje aktivitásának mutációs változásához. Amint itt látható, minden mutáció hatása szubsztrát-specifikus.Annak meghatározása, hogy a szubsztrátok és a mutációk mely kombinációi módosíthatják az enzimatikus aktivitást, a hagyományos in vitro módszerek felhasználásával fáradságos, hangsúlyozva a prediktív virtuális eszközök szükségességét ennek a komplex rejtvénynek a feloldásában.
A személyes és precíziós orvoslás iránti nyilvános és szakmai érdeklődés gyorsan növekszik. Úgy tűnik, hogy a CYP3A4-ben az egyéni polimorfizmus alapján a módosított gyógyszer-anyagcsere előrejelzése csak idő kérdése. Javasoljuk, hogy a különféle etnikai csoportok egyedülálló CYP3A4 SNP-készleteket viseljenek. Valójában az etnikai hovatartozás az első megvalósítható lépés lehet a személyre szabott orvoslásban, megelőzve az egyéni DNS-szűrést mindenki számára. Érdekes módon az etnikum még egy kihatással van a CYP3A4 gyógyszer-anyagcserére, ami fő tényező az ételválasztás és az étkezési szokások meghatározásában. Javasolható, hogy a terápiás rendszert kifejezetten minden etnikai csoport számára meg kell tervezni, legalábbis azokra a gyógyszerekre, amelyeket a CYP3A4 erősen metabolizál. Ez rávilágít az adatbázisok hasznosításának és integrálásának, valamint az alapos tanulás lehetőségeinek annak megállapítására, hogy az SNP-k, az etnikum, az étrendi vegyületek és a gyógyszerek hogyan módosítják a CYP3A4 aktivitását és az orvosi rendszer sikerét. Ebben a tanulmányban a nyilvánosan elérhető adathalmazokat elemezték. Ezek az adatok itt találhatók: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Szerzői közreműködés
Az összes felsorolt szerző jelentős, közvetlen és szellemi hozzájárulást nyújtott a munkát, és jóváhagyta közzétételre.
Összeférhetetlenségi nyilatkozat
A szerzők kijelentik, hogy a kutatást bármilyen kereskedelmi vagy pénzügyi kapcsolat hiányában végezték, amely potenciálisan értelmezhető összeférhetetlenség.
Kiegészítő anyag
S1 ÁBRA | A CYP3A4 3D szalagmodellje és a mutált aminosavak elhelyezkedése a dokkolásra tervezett hét variáns fehérjében. A hem zöld pálcikákként, a Fe2 + vörös gömbként, az in silico elemzésben használt SNP-k vörös szalagként vannak ábrázolva a szalagon, és a mutáns aminosavak R csoportjai a variáns modellekben kifejezetten világosszürke pálcikák. p>
S1 TÁBLÁZAT | CYP3A4 SNP típusok 141 populációban, 456 rokon egyén, 7 etnikai populációt képviselve.
S2. TÁBLÁZAT | CYP3A4 SNP-k etnikai csoportok szerint.
Lábjegyzetek
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov