Inleiding
Het is al decennia lang bekend dat interacties tussen voedsel en geneesmiddelen (FDI) en interacties tussen kruiden en geneesmiddelen het succes van medische behandelingen beperken. Het enorme aantal mogelijke interacties tussen genetische variaties, medische regimes en de talrijke bioactieve verbindingen die in voedsel en kruiden worden aangetroffen, resulteren in een overweldigende complexiteit. Moderne tools zoals big data-analyse, machine learning en simulatie van eiwit-ligand-interacties kunnen ons helpen een hele reeks vragen te beantwoorden: kunnen voedselkeuzes bijdragen aan het mislukken van therapeutische regimes en, zo ja, hoe? Welk (e) voedsel (en) moet (en) worden geconsumeerd voordat een voorgeschreven medicijn wordt ingenomen? En waarschijnlijk de meest opwindende vraag: hoe kunnen we deze tools gebruiken om persoonlijke FDI te voorspellen? Het is duidelijk dat veel antwoorden liggen in het metabolisme van medicijnen, voedsel en kruiden door cytochroom P450 3A4 (CYP3A4) in de lever en het spijsverteringskanaal (Galetin et al., 2010; Basheer en Kerem, 2015).
De meeste genen die coderen voor CYP-enzymen zijn polymorf. Tot op heden is de meest uitgebreide informatiebron die CYP-allelen beschrijft het Pharmacogene Variation Consortium1, waarin minder dan 100 allelen van CYP3A4 zijn vertegenwoordigd. Hiervan zijn minder dan 40 exonische SNP’s (single nucleotide polymorfismen) die resulteren in een gemodificeerde eiwitsequentie. Het kleine aantal proefpersonen in alle eerder gepubliceerde werken over CYP3A4-mutaties levert ons beperkte gegevens op over de werkelijke frequenties van CYP3A4-mutaties in de hele populatie en in gedefinieerde groepen.
Niet alleen die betrouwbare informatie over de incidentie van SNP’s is onvolledig , zijn ook hun klinische implicaties in de meeste gevallen nog onduidelijk (Zanger et al., 2014). Begrijpen welke en wanneer SNP’s klinische betekenis kunnen hebben, is een enorm complexe taak. In vitro assays zijn tijdrovend, duur en praktisch van weinig relevantie gezien het grote aantal mutaties en het oneindige aantal voedsel-medicijncombinaties. Methoden voor moleculaire modellering, waaronder berekeningen van docking en vrije-energiebinding, kunnen dienen om mogelijke effecten van SNP’s en van vele verbindingen op het CYP3A4-gemedieerde metabolisme te voorspellen (Lewis et al., 1998). Niet-covalente, hydrofobe, elektrostatische en van der Waals-interacties dragen bijvoorbeeld allemaal bij aan de oriëntatie van een verbinding en dus aan de binding en reactie ervan op de actieve plaats van een enzym. Deze zullen op hun beurt de affiniteit en specificiteit van het enzym voor verschillende substraten bepalen, en de potentie van enzymremmers (Kirchmair et al., 2012; Basheer et al., 2017).
Hier stellen we een nieuwe voor. benadering voor het meten van de allelfrequentie van CYP3A4-mutaties in verschillende etnische groepen. Deze alomvattende benadering heeft de kracht om mutaties te benadrukken die in bepaalde etnische groepen voorkomen, en in combinatie met screening op chemicaliën die op elkaar inwerken, bijvoorbeeld remmers uit voedsel, zal het mogelijk zijn om de effecten van bepaalde mutaties op de interactie tussen geneesmiddelen en voedsel op te helderen, wat als een eerste dient. stap naar gepersonaliseerde geneeskunde en voeding. Dit werk kan het bewustzijn vergroten van het mogelijke klinische belang van eiwitveranderende CYP3A4-SNP’s en suggereert ook enkele noodzakelijke hulpmiddelen voor de promotie en toepassing van precisie- en gepersonaliseerde geneeskunde.
Materialen en methoden
Database Screening en Data Analyse
De CYP3A4-varianten dataset werd gedownload van de gnomAD browser2 als een CVS-bestand. Python 2.7 met NumPy, panda’s en matplotlib-pakketten werd gebruikt voor gegevensanalyse en visualisatie (zie aanvullend gegevensblad S1). Agglomeratieve hiërarchische clustering werd uitgevoerd met behulp van de Expander 7-software (Shamir et al., 2005) met de Pearson-rangcorrelatiecoëfficiënt als maat voor overeenkomsten en het volledige koppelingstype. Er werd een afstandsdrempel van 0,6 ingesteld voor het groeperen van SNP’s.
In silico Polymorphism Modeling
Maestro 2017-2 release (Schrodinger, New York, NY, Verenigde Staten) werd gebruikt voor de computationele modellering. Het CYP3A4-dockingmodel is gebouwd zoals eerder beschreven (Basheer et al., 2017). Kortom, de CYP3A4-kristalstructuur (PDB-invoer 2V0M) werd verwerkt, gemodificeerd en verfijnd volgens de stappen van de Protein Preparation Wizard. Een koppelingsrooster met een metaalcoördinatiebeperking voor de Fe2 + in de heemgroep werd gegenereerd op basis van het zwaartepunt van ketoconazol in de oorspronkelijke bindingsplaats in de kristalstructuur. Er werden zeven mutaties geselecteerd voor docking-simulaties, één als vertegenwoordiger voor elke etnische groep (tabellen 1, 2). Voor elk variant eiwit werd een enkele puntmutatie geïntroduceerd voorafgaand aan de stappen van de eiwitbereiding. 3D-structuren van liganden werden gegenereerd op basis van 2D-structuren van PubChem3 en voorbereid voor docking met behulp van LigPrep-taak.OPLS3-krachtveld en standaard Glide-opties voor standaardprecisie werden toegepast voor het docking-model, met de uitzondering dat de metalen coördinatiebeperking werd gebruikt, evenals 30 poses voor het aantal poses dat moet worden opgenomen en 10 poses voor het aantal poses om te schrijven uit. Voor elke ligand is het koppelingsresultaat met de laagste Glide-emodel-score geselecteerd.

Tabel 1. Geselecteerde representatieve SNP’s voor zeven etnische groepen.

Tabel 2. Frequentie (%) van geselecteerde mutaties per etnische groep.
Resultaten
De Genome Aggregation Database (gnomAD; zie tekst voetnoot 2) verzamelt zowel exome- als genoomsequencing-gegevens van een breed scala aan grootschalige sequencing-projecten. Het bevat gegevens van 125.748 exoomsequenties en 15.708 sequenties van het hele genoom van 141.456 niet-verwante individuen die zeven etnische populaties vertegenwoordigen (Lek et al., 2016). De GnomAD-database presenteert 856 varianten van CYP3A4, waarvan 397 intronisch en maar liefst 459 exonisch. Van de exonische SNP’s zijn er 312 missense-mutaties, wat aangeeft dat ze de eiwitstructuur beïnvloeden. Het CYP3A4-gen is 34.205 bp lang. De 13 exons omvatten een coderend gebied van 1.512 bp dat een eiwit van 504 aminozuren produceert. De 412 exonische SNP’s met unieke posities in dit gen resulteren in een exonische SNP-dichtheid van 272 / kbp (aanvullende tabel S1).
Berekening van differentiële allelfrequenties per etnische groep laat zien dat sommige populaties hogere frequenties van mutaties vertonen (Figuur 1A). De meeste CYP3A4-mutaties in de Europese bevolking zijn inderdaad zeldzaam, zoals algemeen wordt aangenomen, terwijl mutaties in andere populaties, zoals Afrikaanse en Oost-Aziatische, veel vaker voorkomen (aanvullende tabel S2).
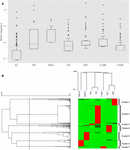
Figuur 1. Analyse van CYP3A4 missense SNP’s in zeven verschillende populaties. (A) Log-schaal boxplot van allelische frequenties. Vakken vertegenwoordigen het interkwartielbereik (IQR), blauwe lijnen vertegenwoordigen de medianen, snorharen vertegenwoordigen gegevens binnen 1,5 IQR en uitschieters worden weergegeven als kleine cirkels. (B) Hiërarchische clustering van allelische frequenties. Elke rij vertegenwoordigt een enkele SNP. Elke kolom vertegenwoordigt een verschillende etnische populatie. De allelfrequentie van de SNP’s in elk van de populaties wordt weergegeven door de kleur van de overeenkomstige cel in het matrixbestand. Groen en rood vertegenwoordigen respectievelijk lage en hoge frequentie. Het bovenste dendrogram vertoont overeenkomsten in het allelfrequentiepatroon tussen elke groep proefpersonen. Het linker dendrogram vertegenwoordigt de clustering van genen in twee groepen. De stippellijn geeft de afstandsdrempel van 0,6 weer die wordt gebruikt voor het splitsen in groepen. EU – Europees (niet-Fins; n = 64.603), FIN – Europees (Fins; n = 12.562), ASH J – Asjkenazisch Joods (n = 5.185), LTN – Latino (n = 17.720), AFR – Afrikaans (n = 12.487), E ASN – Oost-Aziatisch (n = 9.977), S ASN – Zuid-Aziatisch (n = 64.603).
We gebruikten hiërarchische clustering om varianten met vergelijkbare frequentiepatronen te groeperen. Onze data-analyse leverde zeven verschillende clusters op (Figuur 1B). Verder wordt duidelijk waargenomen dat hoogfrequente SNP’s in elk cluster kenmerkend zijn voor één specifieke populatie. Hiërarchische clusteringanalyse van de etnische groepen ondersteunt de associatie tussen genetische variantie en etniciteit door verwante etniciteiten zoals Zuid- en Oost-Aziaten en Finse en niet-Finse Europeanen te groeperen.
Er werd een computermodel gebruikt om te beoordelen de mogelijke invloed van puntmutaties in CYP3A4 op zijn vermogen om substraten en remmers te binden. CYP3A4 kan een breed scala aan endogene en xenobiotische verbindingen oxideren. Hier werd ketoconazol geselecteerd als een representatief medicijn en een zeer efficiënte specifieke remmer; androstenedione en testosteron werden geselecteerd als representatief endogeen hormoon; en demethoxycurcumine en epigallocatechine werden geselecteerd als vertegenwoordigers van bioactieve stoffen in de voeding. Er werd een docking-model gebouwd om de bindingsposities van de geselecteerde verbindingen in de CYP3A4-bindingsplaats te voorspellen. Het model werd eerst gevalideerd door met succes de ketoconazol-pose in de bindingsplaats te herstellen, met een RMSD van 1,52 Å ten opzichte van de oorspronkelijke kristalstructuur. Zeven mutante eiwitten werden ontworpen op basis van de kristalstructuur van het wildtype-eiwit (aanvullende figuur S1). Voor elke etnische groep werd de meest voorkomende unieke mutatie als representatief geselecteerd. Het effect van enkele mutaties op de substraatbinding werd beoordeeld op basis van de vergelijking tussen docking-poses op het natieve eiwit en op variante eiwitten. Veranderingen in koppelingsposities in termen van RMSD worden samengevat in Tabel 3.

Tabel 3. RMSD ten opzichte van de WT van het koppelen van liganden aan bindingsplaatsen van zeven CYP3A4-varianten.
Het effect van CYP3A4 SNP op substraatbinding bleek mutatiesubstraatspecifiek te zijn. Slechts in enkele gevallen veroorzaakten mutaties een verandering in de bindingspositie van een ligand in de bindingspocket. De aanleg van testosteron was hetzelfde in alle zeven geteste varianten. De E262K-, D174H- en K168N-varianten veroorzaakten geen verandering in de bindingspositie in een van de geteste moleculen. De L373F- en T163A-mutaties veranderden echter de bindingshouding van androsteendion zodat het parallel aan de heemgroep werd gepositioneerd in plaats van er loodrecht op, zoals in het WT-eiwit. Ook werd androsteendion geroteerd zodat de cyclopentanongroep proximaal van het heem ligt, in plaats van de cyclohexanongroep in het WT-eiwit. De S222P- en L293P-mutaties veroorzaakten slechts een kleine rotatie in de bindingspositie van androsteendion (Figuur 2A). Van alle onderzochte mutaties veroorzaakte alleen S222P substantiële veranderingen in de koppelingsposities van ketoconazol en demethoxycurcumine op de bindingsplaats (figuren 2B, C); terwijl voor epigallocatechin de pose-veranderende mutatie L373F was (Figuur 2D).
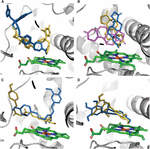
Figuur 2. Modellen van liganden gedokt op de bindingsplaats van CYP3A4. (A) Ketoconazol, (B) androsteendion, (C) demethoxycurcumine en (D) epigallocatechine. De eiwitbindingsplaats wordt weergegeven door grijze linten; heem wordt weergegeven door groene stokjes, koppelingsposities in het WT-eiwit en in S222P- en L373F-mutanten worden respectievelijk weergegeven als oranje, blauwe en violette stokjes. Androstenedione docking-poses in L293P- en in T136A-varianten overlappen de poses in S222P- en in L373F-varianten, respectievelijk.
Discussie
Cytochroom P450 3A4 is het belangrijkste enzym dat verantwoordelijk is voor interacties tussen voedsel en geneesmiddelen. Het huidige onderzoek naar mutaties in CYP3A4 is gericht op enkele tientallen SNP’s die zijn aangetroffen in aangewezen onderzoeken (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al., 2001; ., 2002; Murayama et al., 2002). Zoals hier aangetoond, vormen ze het topje van een ijsberg gezien de prevalentie en mogelijke uitkomsten van CYP3A4-mutaties. De overvloed aan grote genoom- en exoomsequencing-projecten heeft een nieuwe weg geopend voor de identificatie van veel onbekende mutaties. Hier laten we zien dat de eerder gepresenteerde mutaties slechts het topje van de ijsberg zijn, door 856 mutaties aan te tonen die bestaan in CYP3A4, waarvan een derde de eiwitstructuur wijzigt. Met behulp van een cohort van 141.456 niet-verwante individuen werden nauwkeurige allelfrequenties van CYP3A4-mutaties berekend voor zeven afzonderlijke etniciteiten. Voor zover wij weten, is dit de grootste en meest uitgebreide studie met grote gegevens van CYP3A4-exonische mutaties en hun allelfrequenties in verschillende populaties, die tot nu toe is gepubliceerd.
Polymorfe CYP3A4-enzymen kunnen erg belangrijk zijn bij het verklaren verschillen in werkzaamheid en toxiciteit van geneesmiddelen tussen verschillende individuen. Mutaties in het CYP3A4-gen kunnen leiden tot vernietigde, verminderde, veranderde of verhoogde enzymatische activiteit. Exonische mutaties kunnen enzymatische activiteit wijzigen, zoals is aangetoond in enkele klinische onderzoeken met geselecteerde substraten. Enkele gevallen van veranderd metabolisme als gevolg van SNP’s in CYP3A4 zijn al beschreven in de literatuur (Eiselt et al., 2001; Miyazaki et al., 2008). Ondanks het functionele belang en de klinische relevantie van SNP’s in CYP3A4 en mogelijk vanwege hun relatief lage geïdentificeerde frequentie in de algemene populatie, heeft polymorfisme in CYP3A4 niet de aandacht gekregen die het verdient.
Hier dienden zeven mutaties om te voorspellen het effect van SNP’s op substraat- en remmerbindende oriëntatie. In de literatuur verdeelt CYP3A4-polymorfisme de algemene bevolking in drie groepen: slechte metaboliseerders, normale metaboliseerders en snelle metaboliseerders, gebaseerd op intronische SNP’s die expressieniveaus wijzigen in plaats van structuur (Zanger en Schwab, 2013). Onze berekeningen suggereren een aanvullende classificatie: de gewijzigde metaboliseerders. Sommige door ons virtuele model voorgestelde mutaties zouden een verandering in de bindingsoriëntatie van individuele liganden veroorzaken. Deze veranderingen zouden naar verwachting de kans op enzymatische oxidatie verminderen als gevolg van een grotere afstand tot het heem, of leiden tot producten die anders niet zichtbaar zouden zijn tijdens toxiciteitstests die worden uitgevoerd als onderdeel van het ontwikkelingsproces van geneesmiddelen. Zoals ons model echter voorspelt, zijn CYP3A4-mutaties voor de meeste substraten goedaardig.
Gewijzigde positie van een substraat in de bindingspocket als gevolg van structurele verandering van proteïne is slechts één mogelijk mechanisme waardoor een mutatie de activiteit van een proteïne zou kunnen veranderen. . Verminderde verankering van het eiwit aan het membraan, beschadigde substraatleidende kanalen en gecompromitteerde uitgang van de producten presenteren aanvullende mechanismen voor een mutationele verandering in de activiteit van een eiwit. Zoals hier wordt aangetoond, is het effect van elke mutatie substraatspecifiek.Bepalen welke combinaties van substraten en mutaties de enzymatische activiteit zouden kunnen wijzigen, met behulp van traditionele in-vitromethoden, is bewerkelijk, wat de behoefte aan voorspellende virtuele tools benadrukt om deze complexe puzzel op te lossen.
Publieke en professionele interesse in persoonlijke en precisiegeneeskunde groeit snel. Voorspelling van gemodificeerd geneesmiddelmetabolisme op basis van individueel polymorfisme in CYP3A4 lijkt slechts een kwestie van tijd te zijn. Hier stellen we voor dat verschillende etnische groepen unieke sets van CYP3A4 SNP’s dragen. Etniciteit kan inderdaad dienen als een eerste haalbare stap in gepersonaliseerde geneeskunde, voorafgaand aan de implementatie van een individuele DNA-screening voor iedereen. Interessant is dat etniciteit nog een implicatie heeft voor het metabolisme van CYP3A4-geneesmiddelen, omdat het een belangrijke factor is bij het bepalen van voedselkeuzes en voedingsgewoonten. Er kan worden gesuggereerd dat therapeutische regimes specifiek voor elke etnische groep zouden moeten worden ontworpen, in ieder geval voor geneesmiddelen die sterk worden gemetaboliseerd door CYP3A4. Dit benadrukt de mogelijkheden voor het benutten en integreren van databases en diepgaand leren om te identificeren hoe SNP’s, etniciteit, voedingssamenstellingen en medicijnen de CYP3A4-activiteit en het succes van een medisch regime veranderen.
Beschikbaarheid van gegevens
In deze studie zijn openbaar beschikbare datasets geanalyseerd. Deze gegevens zijn hier te vinden: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Auteursbijdragen
Alle vermelde auteurs hebben een substantiële, directe en intellectuele bijdrage geleverd aan de werk, en keurde het goed voor publicatie.
Verklaring inzake belangenconflicten
De auteurs verklaren dat het onderzoek is uitgevoerd in afwezigheid van enige commerciële of financiële relatie die als potentieel kan worden beschouwd belangenconflict.
Aanvullend materiaal
FIGUUR S1 | 3D-lintmodel van CYP3A4 en de locatie van de gemuteerde aminozuren in de zeven variantproteïnen die zijn ontworpen om te koppelen. Heme wordt weergegeven als groene stokjes, Fe2 + wordt weergegeven als een rode bol, SNP’s die worden gebruikt in de in silico-analyse worden weergegeven als rode gebieden op het lint en R-groepen van gemuteerde aminozuren in variantmodellen worden expliciet weergegeven als lichtgrijze stokjes.
TABEL S1 | CYP3A4 SNP-typen in een populatie van 141, 456 niet-verwante individuen die 7 etnische populaties vertegenwoordigen.
TABEL S2 | CYP3A4 SNP’s per etnische groep.
Voetnoten
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov