Introducere
De decenii, se știe că interacțiunile dintre alimente și medicamente (ISD) și interacțiunile dintre plante și medicamente limitează succesul tratamentelor medicale. Numărul enorm de posibile interacțiuni dintre variațiile genetice, regimurile medicale și numeroșii compuși bioactivi găsiți în alimente și ierburi duc la o complexitate copleșitoare. Instrumente moderne precum analiza datelor mari, învățarea automată și simularea interacțiunilor proteină-ligand ne pot ajuta să răspundem la un întreg set de întrebări: alegerile alimentare pot contribui la eșecul regimurilor terapeutice și, dacă da, cum? Ce alimente ar trebui consumate înainte de a lua un medicament prescris? Și probabil cea mai interesantă întrebare: Cum putem folosi aceste instrumente pentru a prezice ISD personale? În mod clar, multe răspunsuri stau în metabolismul medicamentelor, alimentelor și ierburilor de către citocromul P450 3A4 (CYP3A4) în ficat și tractul digestiv (Galetin și colab., 2010; Basheer și Kerem, 2015).
Majoritatea genelor care codifică enzimele CYP sunt polimorfe. Până în prezent, cea mai cuprinzătoare sursă de informații care detaliază alelele CYP este Pharmacogene Variation Consortium1, în care sunt reprezentate mai puțin de 100 de alele ale CYP3A4. Dintre acestea, mai puțin de 40 sunt SNP exonici (polimorfisme cu nucleotide unice) care au ca rezultat o secvență proteică modificată. Numărul mic de subiecți din toate lucrările publicate anterior despre mutațiile CYP3A4 ne oferă date limitate cu privire la frecvențele adevărate ale mutațiilor CYP3A4 în întreaga populație și în grupuri definite.
Nu numai că informațiile fiabile despre incidența SNP sunt incomplete , de asemenea, implicațiile lor clinice sunt încă neclare în majoritatea cazurilor (Zanger și colab., 2014). Înțelegerea care și când SNP-urile ar putea avea semnificație clinică este o sarcină extrem de complexă. Testele in vitro sunt consumatoare de timp, costisitoare și practic cu relevanță redusă, având în vedere cantitatea mare de mutații și numărul nesfârșit de combinații alimente-medicamente. Metodele de modelare moleculară, inclusiv andocarea și calculele de legare a energiei libere, pot servi la prezicerea efectelor potențiale ale SNP-urilor și ale multor compuși asupra metabolismului mediat de CYP3A4 (Lewis și colab., 1998). De exemplu, interacțiunile non-covalente, hidrofobe, electrostatice și van der Waals, toate contribuie la orientarea unui compus și, prin urmare, la legarea și reacția acestuia la locul activ al unei enzime. La rândul lor, acestea vor determina afinitatea și specificitatea enzimei pentru diferite substraturi și potența inhibitorilor enzimei (Kirchmair și colab., 2012; Basheer și colab., 2017).
Aici, propunem un nou abordarea măsurării frecvenței alelice a mutațiilor CYP3A4 în diferite grupuri etnice. Această abordare cuprinzătoare are puterea de a evidenția mutațiile care sunt predominante în anumite grupuri etnice și combinate cu screeningul pentru substanțele chimice care interacționează, de exemplu, inhibitorii din alimente vor permite elucidarea efectelor anumitor mutații asupra interacțiunii medicamente-alimente, servind ca o inițială pas către medicină și nutriție personalizate. Această lucrare poate conștientiza importanța clinică posibilă a SNP-urilor CYP3A4 care modifică proteinele și, de asemenea, sugerează câteva instrumente necesare pentru promovarea și aplicarea medicinii de precizie și personalizate.
Materiale și metode
Screening-ul bazei de date și analiza datelor
Setul de date variante CYP3A4 a fost descărcat din browserul gnomAD2 ca fișier CVS. Python 2.7 cu pachete NumPy, pandas și matplotlib a fost utilizat pentru analiza și vizualizarea datelor (a se vedea fișa de date suplimentară S1). Clusterizarea ierarhică aglomerativă a fost efectuată utilizând software-ul Expander 7 (Shamir și colab., 2005) cu coeficientul de corelație a gradului Pearson ca măsură a asemănărilor și a tipului complet de legătură. Un prag de distanță de 0,6 a fost stabilit pentru gruparea SNP-urilor.
In silico Polymorphism Modeling
Versiunea Maestro 2017-2 (Schrodinger, New York, NY, Statele Unite) a fost utilizată pentru modelare computațională. Modelul de andocare CYP3A4 a fost construit așa cum s-a descris anterior (Basheer și colab., 2017). Pe scurt, structura cristalină CYP3A4 (intrarea PDB 2V0M) a fost procesată, modificată și rafinată urmând pașii Expertului de pregătire a proteinelor. O grilă de andocare cu o constrângere de coordonare a metalului pentru Fe2 + din grupul hem a fost generată pe baza centrului de ketoconazol în situl de legare original din structura cristalină. Șapte mutații au fost selectate pentru simulări de andocare, una ca reprezentant pentru fiecare grup etnic (tabelele 1, 2). Pentru fiecare variantă de proteină, a fost introdusă o mutație cu un singur punct înainte de etapele de preparare a proteinelor. Structurile 3D ale liganzilor au fost generate pe baza structurilor 2D de la PubChem3 și pregătite pentru andocare folosind sarcina LigPrep.Câmpul de forță OPLS3 și opțiunile Glide implicite pentru precizie standard au fost aplicate pentru modelul de andocare, cu excepția faptului că a fost utilizată constrângerea de coordonare a metalului, precum și 30 de poziții pentru numărul de poziții de inclus și 10 poziții pentru numărul de poziții de scris afară. Pentru fiecare ligand, a fost selectat rezultatul de andocare cu cel mai mic scor Glide emodel.

Tabelul 1. SNP-uri reprezentative selectate pentru șapte grupuri etnice.

Tabelul 2. Frecvența (%) mutațiilor selectate în funcție de grup etnic.
Rezultate
Baza de date de agregare a genomului (gnomAD; a se vedea nota de subsol 2) agregează atât date de secvențiere a exomului, cât și date ale genomului dintr-o mare varietate de proiecte de secvențiere pe scară largă. Include date de la 125.748 secvențe de exomi și 15.708 secvențe de genom întreg de la 141.456 indivizi care nu au legătură, reprezentând șapte populații etnice (Lek și colab., 2016). Baza de date GnomAD prezintă 856 de variante ale CYP3A4, dintre care 397 sunt intronice și până la 459 sunt exonice. Dintre SNP-urile exonice, 312 sunt mutații fără sens, indicând faptul că afectează structura proteinelor. Gena CYP3A4 are 34.205 bp lungime. Cei 13 exoni ai săi cuprind o regiune de codificare de 1.512 bp care produce o proteină de 504 aminoacizi. Cele 412 SNP exonice cu poziții unice în această genă au ca rezultat o densitate SNP exonică de 272 / kbp (tabelul suplimentar S1).
Calculul frecvențelor diferențiale ale alelelor pe grup etnic relevă faptul că unele populații prezintă frecvențe mai mari de mutații (Figura 1A). Majoritatea mutațiilor CYP3A4 din populația europeană sunt într-adevăr rare, așa cum se crede în mod obișnuit, în timp ce mutațiile din alte populații, cum ar fi Africa și Asia de Est, sunt mult mai răspândite (Tabelul suplimentar S2).
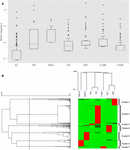
Figura 1. Analiza SNP-urilor CYP3A4 missense în șapte populații distincte. (A) Graficul cutiei la scara jurnal a frecvențelor alelice. Cutiile reprezintă intervalul interquartilei (IQR), liniile albastre reprezintă medianele, mustățile reprezintă date în 1,5 IQR, iar valorile exterioare sunt prezentate ca cercuri mici. (B) Gruparea ierarhică a frecvențelor alelice. Fiecare rând reprezintă un singur SNP. Fiecare coloană reprezintă o populație etnică distinctă. Frecvența de alelă a SNP-urilor din fiecare dintre populații este reprezentată de culoarea celulei corespunzătoare din fișierul matricei. Verde și roșu reprezintă frecvență joasă și respectiv înaltă. Dendrograma superioară prezintă asemănări în tiparul de frecvență alelelor dintre fiecare grup de subiecți. Dendrograma stângă reprezintă gruparea genelor în două grupuri. Linia punctată reprezintă pragul de distanță de 0,6 utilizat pentru împărțirea în grupuri. UE – europeană (non-finlandeză; n = 64.603), FIN – europeană (finlandeză; n = 12.562), ASH J – evreie Ashkenazi (n = 5.185), LTN – latino (n = 17.720), AFR – african (n = 12.487), E ASN – Asia de Est (n = 9.977), S ASN – Asia de Sud (n = 64.603).
Am folosit clustering ierarhic pentru a grupa variante cu modele de frecvență similare. Analiza noastră de date a generat șapte clustere distincte (Figura 1B). Mai mult, se observă în mod clar că SNP-urile de înaltă frecvență din fiecare cluster sunt caracteristice unei populații specifice. Analiza grupării ierarhice a grupurilor etnice sprijină asocierea dintre varianța genetică și etnie prin gruparea etniei conexe, cum ar fi sud-estul și estul asiatic, precum și europenii finlandezi și nefinlandezi.
Un model de calcul a fost utilizat pentru a evalua posibila influență a mutațiilor punctuale în CYP3A4 asupra capacității sale de a lega substraturi și inhibitori. CYP3A4 este capabil să oxideze o gamă largă de compuși endogeni și xenobiotici. Aici, ketoconazolul a fost selectat ca un medicament reprezentativ și un inhibitor specific foarte eficient; androstendiona și testosteronul au fost selectați ca hormoni endogeni reprezentativi; și demetoxicicurcumina și epigalocatechina au fost selectate ca reprezentanți ai bioactivilor dietetici. Un model de andocare a fost construit pentru a prezice pozițiile de legare ale compușilor selectați în situl de legare CYP3A4. Modelul a fost validat mai întâi prin restaurarea cu succes a poziției ketoconazolului în situsul de legare, cu un RMSD de 1,52 Å în raport cu structura cristalină originală. Șapte proteine mutante au fost proiectate pe baza structurii cristaline a proteinei de tip sălbatic (Figura suplimentară S1). Pentru fiecare grup etnic, cea mai frecventă mutație unică a fost selectată ca reprezentativă. Efectul mutațiilor unice asupra legării substratului a fost evaluat pe baza comparației dintre pozițiile de andocare pe proteina nativă și pe proteinele variante. Modificările în pozițiile de andocare în termeni de RMSD sunt rezumate în Tabelul 3.

Tabelul 3. RMSD în raport cu WT-ul liganzilor de andocare la locurile de legare a șapte variante CYP3A4.
S-a constatat că efectul CYP3A4 SNP asupra legării substratului este specific mutației-substrat. Numai în câteva cazuri mutațiile au provocat o modificare a poziției de legare a unui ligand în buzunarul de legare. Poziția de andocare a testosteronului a fost aceeași în toate cele șapte variante testate. Variantele E262K, D174H și K168N nu au provocat o modificare a poziției de legare în niciuna dintre moleculele testate. Cu toate acestea, mutațiile L373F și T163A au schimbat poziția de legare a androstendionei, astfel încât a fost poziționată paralel cu grupul hem, mai degrabă decât perpendicular pe acesta, ca în proteina WT. De asemenea, androstendiona a fost rotită astfel încât gruparea ciclopentanonă să fie localizată în proximitate cu hemul, în locul grupării ciclohexanonă din proteina WT. Mutațiile S222P și L293P au cauzat doar o mică rotație în poziția de legare a androstendionei (Figura 2A). Dintre toate mutațiile examinate, numai S222P a provocat modificări substanțiale în pozițiile de andocare ale ketoconazolului și demetoxicicurcuminei la locul de legare (Figurile 2B, C); întrucât pentru epigallocatechin, mutația care schimbă poziția a fost L373F (Figura 2D).
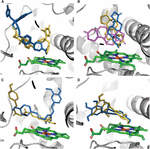
Figura 2. Modele de liganzi andocați la locul de legare al CYP3A4. (A) Ketoconazol, (B) androstendionă, (C) demetoxicicurcumină și (D) epigalocatechină. Situl de legare a proteinelor este reprezentat de panglici cenușii; hemul este reprezentat de bețe verzi, posturi de andocare în proteina WT, iar în mutanții S222P și L373F sunt prezentate sub formă de bastoane portocalii, albastre și, respectiv, violete. Pozițiile de androstendionă de andocare în variantele L293P și T136A se suprapun în ipostazele S222P și, respectiv, în variantele L373F.
Discuție
Citocromul P450 3A4 este enzima principală responsabilă de interacțiunile dintre alimente și medicamente. Cercetările actuale asupra mutațiilor din CYP3A4 s-au concentrat pe câteva zeci de SNP găsite în studii desemnate (Sata și colab., 2000; Dai și colab., 2001; Eiselt și colab., 2001; Hsieh și colab., 2001; Lamba și colab. ., 2002; Murayama și colab., 2002). După cum s-a demonstrat aici, ele reprezintă vârful unui aisberg, având în vedere prevalența și rezultatele potențiale ale mutațiilor CYP3A4. Abundența proiectelor de secvențiere a genomului mare și a exomului a deschis o nouă cale pentru identificarea multor mutații necunoscute. Aici, arătăm că mutațiile prezentate anterior sunt doar vârful aisbergului, demonstrând 856 de mutații existente în CYP3A4, dintre care o treime modifică structura proteinei. Folosind o cohortă de 141.456 indivizi fără legătură, frecvențele alelice precise ale mutațiilor CYP3A4 au fost calculate pentru șapte etnii separate. Din câte știm, acesta este cel mai mare și mai cuprinzător studiu de date mari despre mutațiile exonice CYP3A4 și frecvențele lor de alele în diferite populații, publicat până în prezent.
Enzimele polimorfe CYP3A4 pot fi foarte importante în explicarea diferențe în eficacitatea și toxicitatea medicamentelor între diferiți indivizi. Mutațiile genei CYP3A4 pot duce la desactivarea, reducerea, modificarea sau creșterea activității enzimatice. Mutațiile exonice pot modifica activitatea enzimatică, așa cum s-a demonstrat în câteva studii clinice cu substraturi selectate. Unele cazuri de metabolizare modificată din cauza SNP-urilor în CYP3A4 au fost deja descrise în literatură (Eiselt și colab., 2001; Miyazaki și colab., 2008). În ciuda importanței funcționale și relevanței clinice a SNP-urilor în CYP3A4 și posibil datorită frecvenței relativ scăzute identificate în populația generală, polimorfismul din CYP3A4 nu a primit atenția pe care o merită.
Aici, șapte mutații au servit la prezicerea efectul SNP-urilor asupra orientării de legare a substratului și inhibitorilor. În literatură, polimorfismul CYP3A4 împarte populația generală în trei grupuri – metabolizatori săraci, metabolizatori normali și metabolizatori rapizi, pe baza SNP-urilor intronice care modifică nivelurile de expresie mai degrabă decât structura (Zanger și Schwab, 2013). Calculele noastre sugerează o clasificare suplimentară: metabolizatorii modificați. Unele mutații propuse de modelul nostru virtual ar provoca o schimbare în orientarea de legare a liganzilor individuali. Se așteaptă ca aceste modificări să scadă probabilitatea oxidării enzimatice datorită distanței crescute de hem sau să conducă la produse care altfel nu ar fi evidente în timpul testelor de toxicitate efectuate ca parte a procesului de dezvoltare a medicamentelor. Totuși, așa cum prezice modelul nostru, pentru majoritatea substraturilor mutațiile CYP3A4 sunt benigne.
Poziția modificată a unui substrat în buzunarul de legare datorită modificării structurale a proteinelor este doar un mecanism posibil prin care o mutație ar putea modifica activitatea unei proteine. . Ancorarea afectată a proteinei la membrană, canalele de conducere ale substratului deteriorate și ieșirea compromisă a produselor prezintă mecanisme suplimentare pentru o schimbare mutațională a activității unei proteine. Așa cum se arată aici, efectul fiecărei mutații este specific substratului.Determinarea combinațiilor de substraturi și mutații care ar putea modifica activitatea enzimatică, folosind metode tradiționale in vitro este laborioasă, subliniind necesitatea instrumentelor virtuale predictive în rezolvarea acestui puzzle complex.
Interesul publicului și profesionalului în medicina personală și de precizie crește rapid. Predicția metabolismului modificat al medicamentului pe baza polimorfismului individual în CYP3A4 pare a fi doar o chestiune de timp. Aici, propunem ca grupurile etnice distincte să poarte seturi unice de SNP CYP3A4. Într-adevăr, etnia poate servi ca un prim pas fezabil în medicina personalizată, precedând implementarea unui ecran ADN individual pentru toți. Interesant este că etnia are încă o implicație pentru metabolismul medicamentelor CYP3A4, fiind un factor major în determinarea alegerilor alimentare și a obiceiurilor alimentare. Se poate sugera ca regimurile terapeutice să fie concepute special pentru fiecare grup etnic, cel puțin pentru medicamentele care sunt foarte metabolizate de CYP3A4. Acest lucru evidențiază oportunitățile pentru valorificarea și integrarea bazelor de date și învățarea profundă pentru a identifica modul în care SNP-urile, etnia, compușii dietetici și medicamentele modifică activitatea CYP3A4 și succesul unui regim medical. Seturile de date disponibile publicului au fost analizate în acest studiu. Aceste date pot fi găsite aici: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Contribuțiile autorului
Toți autorii enumerați au adus o contribuție substanțială, directă și intelectuală la și a aprobat-o pentru publicare.
Declarație privind conflictul de interese
Autorii declară că cercetarea a fost efectuată în absența oricărei relații comerciale sau financiare care ar putea fi interpretată ca conflict de interese.
Material suplimentar
FIGURA S1 | Model 3D de panglică al CYP3A4 și localizarea aminoacizilor mutați în cele șapte variante de proteine concepute pentru andocare. Hemul este reprezentat ca niște verzi, Fe2 + este reprezentat ca o sferă roșie, SNP-urile utilizate în analiza in silico sunt reprezentate ca zone roșii pe panglică și grupurile R de aminoacizi mutați în modele variante sunt prezentate în mod explicit ca bastoane gri deschis.
TABEL S1 | Tipuri de SNP CYP3A4 într-o populație de 141, 456 de indivizi care nu au legătură, reprezentând 7 populații etnice.
TABEL S2 | SNP CYP3A4 după grupul etnic.
Note de subsol
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov