Introdução
Por décadas, as interações alimentos-medicamentos (IED) e ervas-medicamentos são conhecidas por limitar o sucesso dos tratamentos médicos. O enorme número de possíveis interações entre variações genéticas, regimes médicos e os numerosos compostos bioativos encontrados em alimentos e ervas resultam em uma complexidade avassaladora. Ferramentas modernas como análise de big-data, aprendizado de máquina e simulação de interações proteína-ligante podem nos ajudar a responder a todo um conjunto de perguntas: As escolhas alimentares podem contribuir para o fracasso dos regimes terapêuticos e, em caso afirmativo, como? Quais alimentos devem ser consumidos antes de tomar um medicamento prescrito? E provavelmente a pergunta mais interessante: como podemos usar essas ferramentas para prever o IED pessoal? Claramente, muitas respostas estão no metabolismo de drogas, alimentos e ervas pelo citocromo P450 3A4 (CYP3A4) no fígado e no trato digestivo (Galetina et al., 2010; Basheer e Kerem, 2015).
A maioria dos genes que codificam as enzimas CYP são polimórficos. Até o momento, a fonte de informações mais abrangente que detalha os alelos CYP é o Pharmacogene Variation Consortium1, no qual menos de 100 alelos do CYP3A4 estão representados. Destes, menos de 40 são SNPs exônicos (polimorfismos de nucleotídeo único) que resultam em uma sequência de proteína modificada. O pequeno número de indivíduos em todos os trabalhos publicados anteriormente sobre mutações de CYP3A4 nos fornece dados limitados sobre as verdadeiras frequências de mutações de CYP3A4 em toda a população e em grupos definidos.
Não só que as informações confiáveis sobre a incidência de SNPs são incompletas , também suas implicações clínicas ainda não são claras na maioria dos casos (Zanger et al., 2014). Entender quais e quando os SNPs podem ter significado clínico é uma tarefa extremamente complexa. Os ensaios in vitro são demorados, caros e praticamente de baixa relevância, considerando a grande quantidade de mutações e o número infinito de combinações de medicamentos e alimentos. Métodos de modelagem molecular, incluindo docking e cálculos de ligação de energia livre, podem servir para prever os efeitos potenciais dos SNPs e de muitos compostos no metabolismo mediado pelo CYP3A4 (Lewis et al., 1998). Por exemplo, interações não covalentes, hidrofóbicas, eletrostáticas e de van der Waals, todas contribuem para a orientação de um composto e, portanto, para sua ligação e reação no sítio ativo de uma enzima. Por sua vez, estes irão determinar a afinidade e especificidade da enzima para diferentes substratos, e a potência dos inibidores da enzima (Kirchmair et al., 2012; Basheer et al., 2017).
Aqui, propomos um novo abordagem para medir a frequência alélica de mutações CYP3A4 em diferentes grupos étnicos. Esta abordagem abrangente tem o poder de destacar mutações que são prevalentes em grupos étnicos específicos e, combinada com a triagem de produtos químicos em interação, por exemplo, inibidores de alimentos permitirão a elucidação dos efeitos de mutações específicas na interação medicamento-alimento, servindo como um ponto inicial passo em direção à medicina e nutrição personalizadas. Este trabalho pode aumentar a conscientização sobre a possível importância clínica dos SNPs de alteração de proteína CYP3A4 e também sugere algumas ferramentas necessárias para a promoção e aplicação de medicina personalizada e de precisão.
Materiais e métodos
Triagem de banco de dados e análise de dados
O conjunto de dados de variantes CYP3A4 foi baixado do navegador gnomAD2 como um arquivo CVS. Python 2.7 com pacotes NumPy, pandas e matplotlib foi usado para análise e visualização de dados (consulte a Folha de Dados Suplementar S1). O agrupamento hierárquico aglomerativo foi realizado usando o software Expander 7 (Shamir et al., 2005) com o coeficiente de correlação de Pearson como medida de semelhanças e tipo de ligação completa. Um limite de distância de 0,6 foi definido para o agrupamento de SNPs.
Modelagem de polimorfismo in silico
A versão Maestro 2017-2 (Schrodinger, New York, NY, Estados Unidos) foi usada para o modelagem computacional. O modelo de docking CYP3A4 foi construído conforme descrito anteriormente (Basheer et al., 2017). Resumidamente, a estrutura cristalina do CYP3A4 (entrada 2V0M do PDB) foi processada, modificada e refinada seguindo as etapas do Assistente de Preparação de Proteínas. Uma grade de acoplamento com uma restrição de coordenação de metal para o Fe2 + no grupo heme foi gerada com base no centróide do cetoconazol no local de ligação original na estrutura cristalina. Sete mutações foram selecionadas para simulações de docking, uma como representante de cada grupo étnico (Tabelas 1, 2). Para cada proteína variante, uma única mutação pontual foi introduzida antes das etapas de preparação da proteína. Estruturas 3D de ligantes foram geradas com base em estruturas 2D do PubChem3 e preparadas para docking usando a tarefa LigPrep.OPLS3 campo de força e opções de deslizamento padrão para precisão padrão foram aplicadas para o modelo de encaixe, com a exceção de que a restrição de coordenação de metal foi usada, bem como 30 poses para o número de poses a incluir e 10 poses para o número de poses a escrever Fora. Para cada ligante, o resultado de docking com a menor pontuação do emodelo Glide foi selecionado.

Tabela 1. SNPs representativos selecionados para sete grupos étnicos.

Tabela 2. Frequência (%) das mutações selecionadas por grupo étnico.
Resultados
O banco de dados de agregação de genoma (gnomAD; consulte a nota de rodapé do texto 2) agrega dados de sequenciamento de exoma e genoma de uma ampla variedade de projetos de sequenciamento em grande escala. Inclui dados de 125.748 sequências de exoma e 15.708 sequências de genoma completo de 141.456 indivíduos não relacionados, representando sete populações étnicas (Lek et al., 2016). O banco de dados GnomAD apresenta 856 variantes do CYP3A4, das quais 397 são intrônicas e até 459 são exônicas. Dos SNPs exônicos, 312 são mutações missense, indicando que afetam a estrutura da proteína. O gene CYP3A4 tem 34.205 bp de comprimento. Seus 13 exons compreendem uma região codificadora de 1.512 bp que produz uma proteína de 504 aminoácidos. Os 412 SNPs exônicos com posições únicas neste gene resultam em uma densidade SNP exônica de 272 / kbp (Tabela Suplementar S1).
O cálculo das frequências diferenciais de alelos por grupo étnico revela que algumas populações exibem frequências mais altas de mutações (Figura 1A). A maioria das mutações do CYP3A4 na população europeia são realmente raras, como comumente se pensa, enquanto as mutações em outras populações, como na África e no Leste Asiático, são muito mais prevalentes (Tabela Suplementar S2).
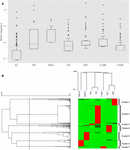
Figura 1. Análise de SNPs missense CYP3A4 em sete populações distintas. (A) Gráfico de caixa da escala logarítmica das frequências alélicas. As caixas representam o intervalo interquartil (IQR), as linhas azuis representam as medianas, os bigodes representam os dados dentro de 1,5 IQR e os outliers são apresentados como pequenos círculos. (B) Agrupamento hierárquico de frequências alélicas. Cada linha representa um único SNP. Cada coluna representa uma população étnica distinta. A frequência do alelo dos SNPs em cada uma das populações é representada pela cor da célula correspondente no arquivo de matriz. Verde e vermelho representam baixa e alta frequência, respectivamente. O dendrograma superior mostra semelhanças no padrão de frequência do alelo entre cada grupo de indivíduos. O dendrograma esquerdo representa o agrupamento de genes em dois grupos. A linha tracejada representa o limite de distância de 0,6 usado para dividir em grupos. EU – europeu (não finlandês; n = 64.603), FIN – europeu (finlandês; n = 12.562), ASH J – judeu Ashkenazi (n = 5.185), LTN – latino (n = 17.720), AFR – africano (n = 12.487), E ASN – Leste Asiático (n = 9.977), S ASN – Sul da Ásia (n = 64.603).
Usamos agrupamento hierárquico para agrupar variantes com padrões de frequência semelhantes. Nossa análise de dados resultou em sete grupos distintos (Figura 1B). Além disso, é claramente observado que os SNPs de alta frequência em cada cluster são característicos de uma população específica. A análise de agrupamento hierárquico dos grupos étnicos suporta a associação entre variância genética e etnia agrupando etnias relacionadas, como asiáticos do sul e do leste, bem como europeus finlandeses e não finlandeses.
Um modelo computacional foi usado para avaliar a possível influência de mutações pontuais no CYP3A4 na sua capacidade de se ligar a substratos e inibidores. O CYP3A4 é capaz de oxidar uma ampla gama de compostos endógenos e xenobióticos. Aqui, o cetoconazol foi selecionado como uma droga representativa e um inibidor específico muito eficiente; androstenediona e testosterona foram selecionadas como hormônio endógeno representativo; e desmetoxicurcumina e epigalocatequina foram selecionadas como representantes dos bioativos da dieta. Um modelo de docking foi construído para prever as posições de ligação dos compostos selecionados no local de ligação do CYP3A4. O modelo foi validado pela primeira vez restaurando com sucesso a postura do cetoconazol no sítio de ligação, com um RMSD de 1,52 Å em relação à estrutura cristalina original. Sete proteínas mutantes foram projetadas com base na estrutura cristalina da proteína de tipo selvagem (Figura Suplementar S1). Para cada grupo étnico, a mutação única mais frequente foi selecionada como representativa. O efeito de mutações únicas na ligação do substrato foi avaliado com base na comparação entre as posições de encaixe na proteína nativa e nas proteínas variantes. As mudanças nas poses de encaixe em termos de RMSD estão resumidas na Tabela 3.

Tabela 3. RMSD em relação ao WT de ligantes de docking a locais de ligação de sete variantes de CYP3A4.
O efeito do SNP do CYP3A4 na ligação do substrato foi considerado específico para mutação do substrato. Apenas em alguns casos, as mutações causaram uma mudança na postura de ligação de um ligante na bolsa de ligação. A pose de encaixe da testosterona foi a mesma em todas as sete variantes testadas. As variantes E262K, D174H e K168N não causaram uma alteração na postura de ligação em nenhuma das moléculas testadas. No entanto, as mutações L373F e T163A mudaram a postura de ligação da androstenediona de modo que ela foi posicionada paralela ao grupo heme em vez de perpendicular a ele, como na proteína WT. Além disso, a androstenediona foi girada de modo que o grupo ciclopentanona esteja localizado próximo ao heme, em vez do grupo ciclohexanona na proteína WT. As mutações S222P e L293P causaram apenas uma pequena rotação na postura de ligação da androstenediona (Figura 2A). De todas as mutações examinadas, apenas S222P causou alterações substanciais nas posições de acoplamento do cetoconazol e da demetoxicurcumina no local de ligação (Figuras 2B, C); enquanto para epigalocatequina, a mutação de mudança de pose foi L373F (Figura 2D).
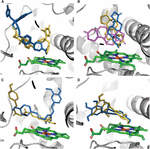
Figura 2. Modelos de ligantes ancorados no local de ligação do CYP3A4. (A) Cetoconazol, (B) androstenediona, (C) desmetoxicurcumina e (D) epigalocatequina. O local de ligação da proteína é representado por fitas cinza; heme é representado por bastões verdes, as posições de encaixe na proteína WT e nos mutantes S222P e L373F são mostrados como bastões laranja, azul e violeta, respectivamente. As poses de acoplamento da androstenediona nas variantes L293P e T136A se sobrepõem às poses nas variantes S222P e L373F, respectivamente.
Discussão
O citocromo P450 3A4 é a principal enzima responsável pelas interações alimentos-medicamentos. A pesquisa atual sobre mutações no CYP3A4 tem se concentrado em algumas dezenas de SNPs encontrados em estudos designados (Sata et al., 2000; Dai et al., 2001; Eiselt et al., 2001; Hsieh et al., 2001; Lamba et al. ., 2002; Murayama et al., 2002). Conforme demonstrado aqui, eles representam a ponta do iceberg, considerando a prevalência e os resultados potenciais das mutações do CYP3A4. A abundância de projetos de sequenciamento de genomas grandes e exomas abriu um novo caminho para a identificação de muitas mutações desconhecidas. Aqui, mostramos que as mutações apresentadas anteriormente são apenas a ponta do iceberg, ao demonstrar 856 mutações existentes no CYP3A4, das quais um terço modifica a estrutura da proteína. Usando uma coorte de 141.456 indivíduos não aparentados, as frequências alélicas precisas de mutações CYP3A4 foram calculadas para sete etnias diferentes. Até onde sabemos, este é o maior e mais abrangente estudo de grandes dados de mutações exônicas de CYP3A4 e suas frequências de alelos em diferentes populações, publicado até o momento.
Enzimas polimórficas de CYP3A4 podem ser muito importantes para explicar diferenças na eficácia e toxicidade do medicamento entre diferentes indivíduos. Mutações no gene CYP3A4 podem levar à abolição, redução, alteração ou aumento da atividade enzimática. Mutações exônicas podem modificar a atividade enzimática, como foi demonstrado em alguns estudos clínicos com substratos selecionados. Alguns casos de metabolismo alterado devido a SNPs no CYP3A4 já foram descritos na literatura (Eiselt et al., 2001; Miyazaki et al., 2008). Apesar da importância funcional e relevância clínica dos SNPs no CYP3A4 e possivelmente devido à sua frequência identificada relativamente baixa na população em geral, o polimorfismo no CYP3A4 não recebeu a atenção que merece.
Aqui, sete mutações serviram para prever o efeito dos SNPs na orientação de ligação ao substrato e ao inibidor. Na literatura, o polimorfismo CYP3A4 divide a população geral em três grupos – metabolizadores fracos, metabolizadores normais e metabolizadores rápidos, com base em SNPs intrônicos que modificam os níveis de expressão em vez da estrutura (Zanger e Schwab, 2013). Nossos cálculos sugerem uma classificação adicional: os metabolizadores alterados. Algumas mutações propostas por nosso modelo virtual causariam uma mudança na orientação de ligação de ligantes individuais. Espera-se que essas alterações diminuam a probabilidade de oxidação enzimática devido ao aumento da distância do heme ou levem a produtos que, de outra forma, não seriam evidentes durante os testes de toxicidade realizados como parte do processo de desenvolvimento do medicamento. No entanto, como nosso modelo prevê, para a maioria dos substratos, as mutações no CYP3A4 são benignas.
A posição modificada de um substrato no bolso de ligação devido à mudança estrutural da proteína é apenas um mecanismo possível pelo qual uma mutação pode alterar a atividade de uma proteína . Ancoragem prejudicada da proteína à membrana, canais principais de substrato danificados e saída comprometida dos produtos apresentam mecanismos adicionais para uma mudança mutacional na atividade de uma proteína. Conforme mostrado aqui, o efeito de cada mutação é específico do substrato.Determinar quais combinações de substratos e mutações podem modificar a atividade enzimática, usando métodos tradicionais in vitro é trabalhoso, enfatizando a necessidade de ferramentas virtuais preditivas para resolver esse quebra-cabeça complexo.
Interesse público e profissional na medicina pessoal e de precisão está crescendo rapidamente. A previsão do metabolismo do medicamento modificado com base no polimorfismo individual no CYP3A4 parece ser apenas uma questão de tempo. Aqui, propomos que grupos étnicos distintos apresentem conjuntos únicos de SNPs CYP3A4. Na verdade, a etnia pode servir como uma primeira etapa viável na medicina personalizada, precedendo a implementação de uma triagem de DNA individual para todos. Curiosamente, a etnia tem mais uma implicação para o metabolismo do medicamento CYP3A4, sendo um fator importante na determinação das escolhas alimentares e hábitos alimentares. Pode-se sugerir que os regimes terapêuticos devem ser projetados especificamente para cada grupo étnico, pelo menos para medicamentos que são altamente metabolizados pelo CYP3A4. Isso destaca as oportunidades de aproveitar e integrar bancos de dados e aprendizado profundo para identificar como SNPs, etnia, compostos dietéticos e medicamentos modificam a atividade do CYP3A4 e o sucesso de um regime médico.
Disponibilidade de dados
Conjuntos de dados disponíveis publicamente foram analisados neste estudo. Esses dados podem ser encontrados aqui: http://gnomad.broadinstitute.org/gene/ENSG00000160868.
Contribuições dos autores
Todos os autores listados fizeram uma contribuição substancial, direta e intelectual para o trabalho e aprovou-o para publicação.
Declaração de conflito de interesses
Os autores declaram que a pesquisa foi conduzida na ausência de quaisquer relações comerciais ou financeiras que pudessem ser interpretadas como um potencial conflito de interesses.
Material Complementar
FIGURA S1 | Modelo de fita 3D do CYP3A4 e a localização dos aminoácidos mutantes nas sete proteínas variantes projetadas para encaixe. Heme é representado como bastões verdes, Fe2 + é representado como uma esfera vermelha, SNPs usados na análise in silico são representados como áreas vermelhas na fita e grupos R de aminoácidos mutados em modelos variantes são mostrados explicitamente como bastões cinza claro.
TABELA S1 | Tipos de SNP CYP3A4 em uma população de 141.456 indivíduos não relacionados, representando 7 populações étnicas.
TABELA S2 | CYP3A4 SNPs por grupo étnico.
Notas de rodapé
- ^ www.pharmvar.org
- ^ https://gnomad.broadinstitute.org
- ^ https://pubchem.ncbi.nlm.nih.gov